 Bioanorganische Chemie (AC6)
Bioanorganische Chemie (AC6)  Bioanorganische Chemie (AC6)
Bioanorganische Chemie (AC6) 2 SWS, Nächster Termin: SS 2016.
Bitte beachten Sie: Drucken Sie bitte nicht das Skript aus. In der ersten Vorlesungsstunde erfahren Sie, wie Sie an ein aktuelles Skript kommen. Das alte Skript steht noch hier, damit Sie eine Idee bekommen, worum es im Prinzip geht.
Alte Klausuren:
| SS 2015: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2011: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2010: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2009: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2008: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2007: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| 3. Klausur | Lösung | |
| SS 2006: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2005: | Klausur | Lösung |
| 2. Klausur | Lösung | |
| SS 2004: | Klausur | Lösung |
Ergebnis der Klausur vom 7. Oktober 2015. (Notenschlüssel.)
Bachelorstudiengang Chemie und Biochemie: Studierende im 6. Semester. Die Vorlesung ist so aufgebaut, dass komplexchemische Konzepte in ihrer Bedeutung für biochemische Vorgänge dargestellt werden. Sie können sich hier testen, ob Sie die Grundlagen verstanden haben.
Die Vorlesung Bioanorganische Chemie fragt in knapp 20 Abschnitten danach, wie die Natur wesentliche Prinzipien der Koordinationschemie umsetzt. Als „Aufhänger“ dient jeweils das aktive Zentrum eines Enzyms. Zuerst wird der biochemische Zusammenhang knapp dargestellt, im Mittelpunkt steht dann der koordinationschemische Sachverhalt, dessen Diskussion oft ein Gefühl dafür vermittelt, warum die Natur im Verlauf der Evolution das ausgewählte katalytische Problem so und nicht anders gelöst hat.
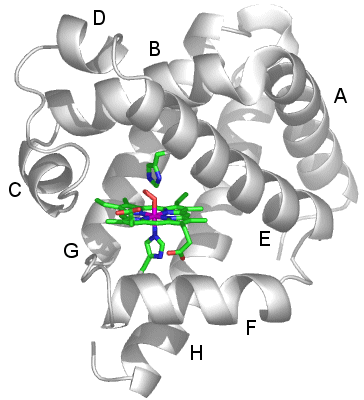
Neben der bei jedem Thema angegebenen Literatur wurde als Quelle für die Enzymstrukturen die PDB, die Protein Data Bank genutzt (http://www.rcsb.org/pdb/), von der die Ergebnisse von Beugungsexperimenten und NMR-spektroskopischen Untersuchungen heruntergeladen werden können. Unter diesem Link findet sich auch eine Fülle frei verfügbarer Programme für die graphische Darstellung der Strukturen. Darunter findet sich auch Pymol (http://pymol.org), mit dem alle Abbildungen in diesem Skript angefertigt wurden.
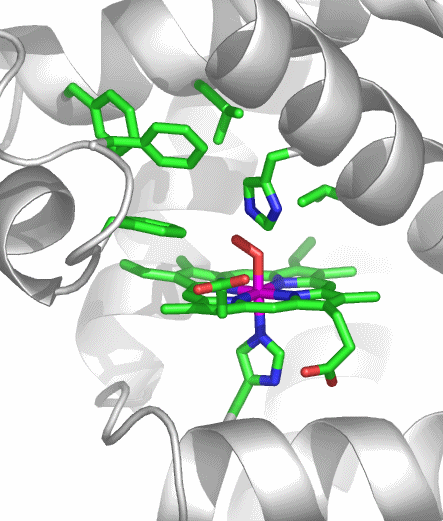
In allen Abbildungen wurde der nicht zum aktiven Zentrum gehörende Teil des Enzyms in der üblichen grob schematischen Weise dargestellt, bei der alle Aminosäureseitenketten weggelassen sind und bei der Hauptkette lediglich α-Helices und β-Faltblattabschnitte hervorgehoben sind. Das aktive Zentrum ist dagegen in Stab-Darstellung gezeichnet. Die Farben der einzelnen Atome sind einheitlich gewählt: Kohlenstoff grün, Stickstoff blau, Sauerstoff rot, Schwefel orange und das erste Metall violett. Gezeigt ist meist sowohl das vollständige Enzym (Holoenzym) als auch das aktive Zentrum in Vergrößerung, oft in derselben Ausrichtung. Ist das aktive Zentrum bereits in der Holoenzym-Darstellung gut sichtbar, wurde auf die Vergrößerung verzichtet.
Man beachte bei der Betrachtung von Struktureinzelheiten die methodischen Beschränkungen der Röntgenstrukturanalyse. Die Positionen der streuschwachen Wasserstoffatome sind in aller Regel nicht direkt bestimmbar, damit wird auch der Protonierungsgrad funktioneller Gruppen nicht erhalten. Ob eine einem einzelnen Sauerstoffatom zugeordnete Elektronendichte in der Nähe eines Metallzentrums einen Oxido-, einen Hydroxido- oder einen Aqua-Ligand darstellt, bleibt also offen. Erscheinen in einer Abbildung Wasserstoffatome, so hat dies mehr illustrativen Charakter.
Prüfen Sie hier, ob Ihr Browser das Skript korrekt darstellt.
2005 waren ca. 300 Enzyme strukturell aufgeklärt. Eine Strukturanalyse in atomarer Auflösung bietet den notwendigen Rahmen zur Analyse des Reaktionsverlaufs im aktiven Zentrum des Enzyms. Ebenso wie in der technischen Katalyse finden sich auch in den aktiven Zentren von Enzymen Metallzentren als Ort der katalytischen Umsetzung des jeweiligen Substrats. Die besonders reiche Chemie von Metallen – vor allem von Übergangsmetallen – ist die offensichtliche Ursache dafür, dass Metalle in den aktiven Zentren von Enzymen von viel größerer Bedeutung sind als anderswo in der Biochemie: etwa die Hälfte der strukturell aufgeklärten Enzyme sind metallhaltige Proteine! [cu2] (Nach einer anderen Quelle sind ein Drittel aller Enzyme Metalloenzyme (A. C Rosenzweig, D. M. Dooley, Current Opinion in Chemical Biology 2006, 10, 89–90).
Die inzwischen soliden Kenntnisse zum Ablauf einer biochemischen Katalyse sind jedoch nicht nur der enormen Entwicklung der Strukturbiologie in den letzten beiden Jahrzehnten zu verdanken. Das statische Bild, das eine Strukturanalyse vermittelt, enthält keine unmittelbare Information über den Ablauf einer Reaktion. Neben weiteren experimentellen Methoden wie der gerichteten Mutation und der spektroskopischen Untersuchung sind es vor allem die Erfolge der Computerchemie, die viel zu einer detaillierten Analyse des Reaktionsgeschehens beitragen.
Die Parallele hinsichtlich der Bedeutung von Übergangsmetallen sowohl bei der technischen als auch bei der biologischen Katalyse ist unübersehbar. Gemeinsame Grundlage beider Disziplinen sind die Regeln der Koordinationschemie, die sich in der biochemischen Katalyse auf hohem Niveau studieren lassen, nämlich optimiert im Verlauf einer ca. 2 Milliarden Jahre dauernden Evolution. Jeder, der katalytische Reaktionen entwerfen oder optimieren will, findet daher beim Studium metallhaltiger Enzyme Lehrbeispiele vor. Es ist die Aufgabe und der Reiz der Bioanorganischen Chemie, die biochemischen Befunde in verallgemeinerbare koordinationschemische Regeln zu fassen, die eine Übertragung des Vorbildes der Natur auf Probleme der Synthesechemie erlauben.
E. I. Solomon, R. K. Szilagyi, S. DeBeer George, L. Basumallick: Electronic Structures of Metal Sites in Proteins and Models: Contributions to Function in Blue Copper Proteins. Chem. Rev. 2004, 104, 419–458[cu2].
Die Acidität der schwachen Brønsted-Säure Wasser wird durch Koordination an die Lewis-Säure Zn2+ erhöht. Die konjugierte Base OH− steht im aktiven Zentrum des Enzyms in hoher Konzentration als Nukleophil zur Verfügung. Einer denkbaren Erniedrigung der Nukleophilie durch die Bindung an die Lewis-Säure steht eine hohe Konzentration an Nukleophil gegenüber. Modellverbindungen zeigen, dass die Lewis-Acidität von Zink(II) mit der Koordinationszahl feinabgestimmt werden kann: ein erstes Beispiel für die Bedeutung der Modellierung zum Verständnis einer katalytischen Reaktion.
Carboanhydrase gehört sowohl von der Verbreitung im Tier- und Pflanzenreich als auch von der biochemischen Bedeutung her zu den wichtigsten Enzymen überhaupt. CA war das erste von heute ca. 200 bekannten Enzymen, die als Zinkenzyme erkannt wurden. (Der menschliche Körper enthält ca. 2 g Zink, das damit hinter Eisen das mengenmäßig zweitwichtigste Metall ist.) Die von CA katalysierte Reaktion mutet fast primitiv an:
CO2 + 2 H2O ⇄ HCO3− + H3O+
Es handelt sich also „nur“ um die Vorbereitung einer Gleichgewichtseinstellung zwischen Lösung und Gasraum, bei uns Menschen zwischen dem Hydrogencarbonat des Blutplasmas und dem Kohlendioxid in den Lungenbläschen. Dass dieser einfache Vorgang kinetisch gehemmt ist und der Katalyse bedarf, erkennt man spätestens dann, wenn man im Biergarten vor einer frisch gezapften Mass sitzt. Auch nach längerer Zeit „bitzelt“ ein Schluck auf der Zunge. Es wird also noch Kohlensäure freigesetzt, die (Gott sei Dank) eben nicht in den ersten Sekunden nach dem Zapfen die wässrige Phase verlassen hat, um so das thermodynamische Gleichgewicht einzustellen – wirksames Veratmen von CO2 ist unkatalysiert also offensichtlich nicht möglich.
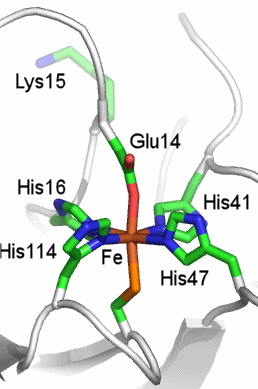
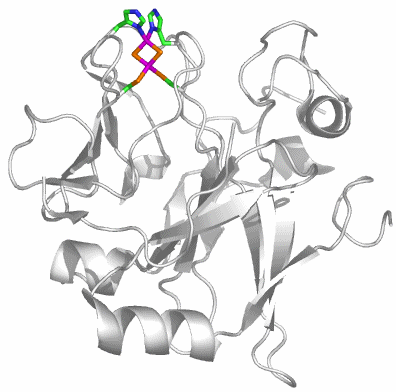
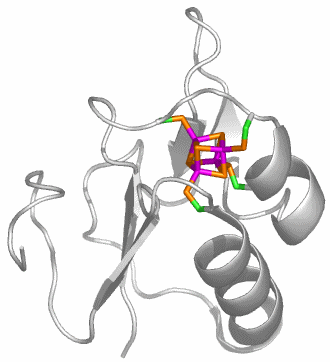
CA besteht aus einem einzelnen Proteinstrang von ca. 260 Aminosäuren. Es sind mehr als 400 Strukturanalysen an CAs und CA-Hemmstoff-Komplexen in der PDB hinterlegt (abfragen unter carbonic anhydrase). Die hier abgebildete Molekülstruktur wurde bei einer Röntgenstrukturanalyse an Kristallen von humaner CA II in einer Auflösung von 2 Å erhalten (PDB-Eintrag: 1CA2, eine mit 0.9 Å hochaufgelöste Struktur wird in [ca_highres_2010] beschrieben).
Holoenzym:
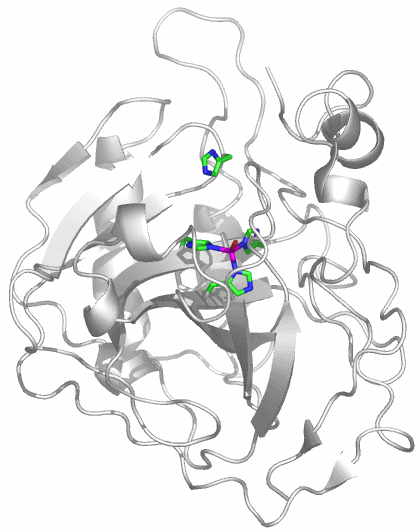
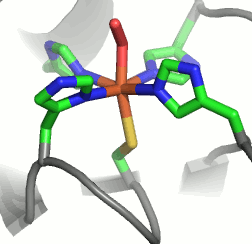
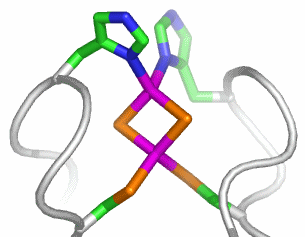
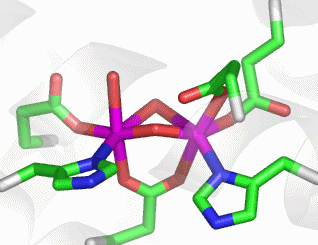
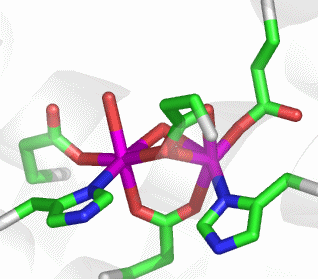
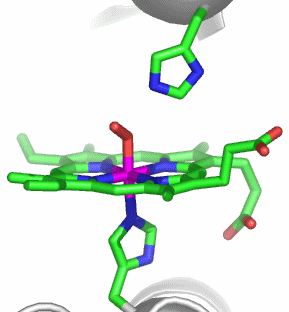
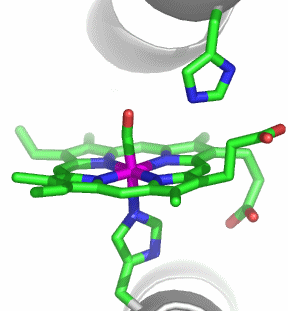
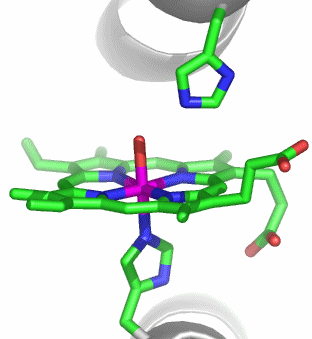
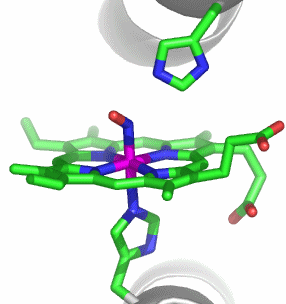
Im aktiven Zentrum binden drei Histidin-Reste ein vierfach koordiniertes Zink-Ion. Die vierte Koordinationsstelle wird von einem Wasser/Hydroxido-Ligand belegt. Unter den Aminosäure-Seitenketten in der näheren Umgebung des aktiven Zentrums – im Bild oberhalb der Zn(His)3-Einheit – wird der Histidin-64-Rest in der Rolle eines „proton shuttles“ gesehen:
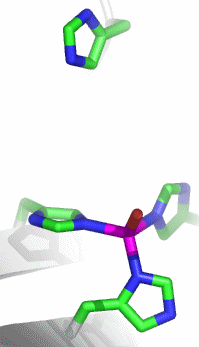
Die Ladung des gezeigten Ausschnitts ist am Metall lokalisiert.
Der Cyclus ist in der literaturüblichen Weise als Umwandlung von CO2 in Hydrogencarbonat dargestellt, wie er bei der CO2-Aufnahme durch photosynthetisierende grüne Pflanzen abläuft. Man beachte, dass er bei der Ausscheidung von CO2 im Gegenuhrzeigersinn abläuft. Der Cyclus beginnt mit dem Enzym in der Ruheform, bei der entsprechend dem pKA-Wert des Aqua-Liganden von ca. 7 dieser vor allem in der Hydroxido-Form vorliegt (pH-Wert des Blutplasmas: 7.4). Das vom Aqua-Liganden abgespaltene Proton wird von His64 gebunden. Da es im weiteren Verlauf der Katalyse wieder in den Kreislauf zurückfließt, dient His64 als „proton shuttle“.
Man beachte, dass eine wichtige Einzelheit einer wirksamen Katalyse darin besteht, dass alle während der Reaktion bewegten Fragmente einen definierten Bindungspartner vorfinden. Es ist also keineswegs sekundär, dass das Proton nicht in die Umgebung entlassen wird und dieser bei Bedarf wieder entzogen wird. Der Hydroxido-Ligand ist das eigentliche Agens, das nun das Elektrophil CO2 angreift. Im nächsten Schritt entsteht ein Hydrogencarbonato-Ligand. Die blau eingezeichneten Pfeile entsprechen den Vorstellungen, die in der Literatur als „Lindskog-Mechanismus“ bezeichnet werden. Dieser hat in den letzten Jahren bei computerchemischen Rechnungen gegenüber einem konkurrierenden, hier nicht diskutierten „Lipscomb-Mechanismus“ an Wahrscheinlichkeit gewonnen. Die Formulierung der Kohlensäure im letzten Reaktionsschritt als Hydrogencarbonat spiegelt deren pKA-Wert von 6.5 wider.
Eine kurze Anmerkung zur oben geschilderten “Biergartenbeobachtung”: wenn Sie dann endlich das Bier nicht weiter stehenlassen und einen Schluck nehmen, dann prickelt es auf Ihrer Zunge durch die Wirkung einer dort lokalisierten Carboanhydrase IV. An dieser Stelle würde ein Carboanhydrase-Hemmer das Vergnügen trüben [ca_2010].
Viele andere Zinkenzyme dienen der Hydrolyse polarer Bindungen. So enthalten Proteasen und Esterasen oft Zink in ihrem aktiven Zentrum. Das Reaktionsprinzip ist das gleiche wie bei CA. Das eigentliche Agens ist ein Hydroxido-Ligand, der als Nukleophil das Kohlenstoffatom polarer C-N- oder C-O-Bindungen angreift.
Die Diskussion der biochemischen Daten konzentriert sich auf die Frage: Was ist eigentlich das Besondere an CA und hydrolytischen Zinkenzymen? Entwickelt die Natur einen aufwendigen Liganden – nichts anderes ist das Apoenzym – für eine Reaktion, die man im Labor mit einer Prise Zinksulfat erledigen könnte? Wir reden also über Modellierung. Wichtige Daten hierzu sind in Fig. 1 von [ca_1999] zusammengestellt, nämlich pKA-Werte synthetischer Zinkkomplexe: Aqua-{N,N- bis(2-picolyl)-N-carboxymethyl-amin}-imidazol-zink (Koordinationszahl 6, pKA 9.5); Aqua-{N,N,N-tris(benzimidazolylmethyl)amin-zink (Koordinationszahl 5, pKA 8.0); Aqua-{hydrido-tris(tert-butyl-pyrazolyl)borato}-zink (Koordinationszahl 4, pKA 6.5). Einen Überblick über das Zusammenspiel Modellierung–Enzymeigenschaften gibt [ca_2007]. Einen umfassenderen Überblick erhalten Sie in [ca_2004].
Bei der Klausur sollten Sie eine Idee haben zu: Aufbau und Acidität von Aqua-Metall-Komplexen; Faktoren, welche die Acidität von metallgebundenen Aqua-Liganden beeinflussen (Koordinationszahl, Ladungsdichte am Zentralmetall, Einbindung der Protonen in Wasserstoffbrückenbindungen).
[ca_highres_2010]
B. S. Avvaru, C. U. Kim, K. H. Sippel, S. M. Gruner, M. Agbandje-McKenna, D. N. Silverman, R. McKenna:
A Short, Strong Hydrogen Bond in the Active Site of Human Carbonic Anhydrase II.
Biochemistry 2010, 49, 249–251.
doi:
10.1021/bi902007b
[ca_2010]
A. Dunkel, T. Hofmann:
Carboanhydrase IV vermittelt das Prickeln der Kohlensäure in Getränken.
Angew. Chem. 2010, 122, 3037–3039.
doi:
10.1002/ange.200906978
[ca_2007]
H. Vahrenkamp:
Why does nature use zinc – a personal view.
Dalton Trans. 2007, 4751–4759.
doi:
10.1039/b712138e
[ca_2004]
G. Parkin:
Synthetic Analogues Relevant to the Structure and Function of Zinc Enzymes.
Chem. Rev. 2004, 104, 699–768.
doi:
10.1021/cr0206263
[ca_1999]
H. Vahrenkamp:
Transitions, Transition States, Transition State Analogues: Zinc Pyrazolylborate Chemistry Related to Zinc Enzymes.
Acc. Chem. Res. 1999, 32, 589–596.
doi:
10.1021/ar9703185
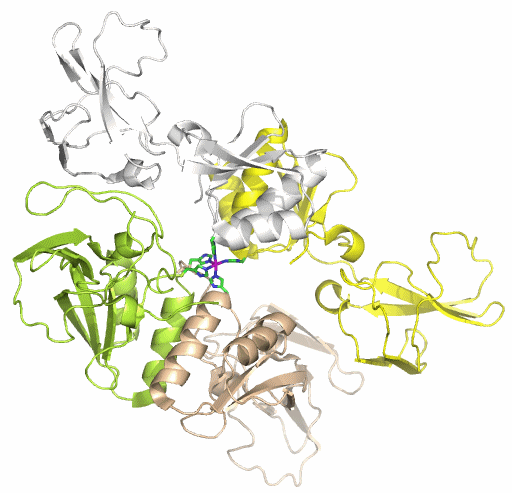
Am Beispiel eines zweiten Säure/Base-Katalysators – Urease – wird eine wichtige Variable vorgestellt, welche die Natur zum Aufbau eines katalytischen Zentrums nutzt: die Nuklearität, also die Zahl der Metallatome im aktiven Zentrum. Bei der Urease spielen bei der Fixierung des Substrats und der katalytischen Transformation zwei Metallzentren zusammen. Die Formulierung eines zutreffenden Katalysecyclus hat bei der Urease einen handfesten technischen Hintergrund, nämlich die Entwicklung wirksamer Urease-Hemmstoffe. Für diese gibt es Bedarf, da ein Großteil des wichtigsten Stickstoffdüngers Harnstoff durch Urease von Bodenbakterien zersetzt wird. Bei Urease fragen wir auch nach der Biosynthese von Metallzentren. Sie werden lernen, dass reaktive Metall-Ionen wie eben Nickel, aber auch Kupfer, Eisen, etc., keineswegs als hydratisierte Ionen im Zellinneren herumschwimmen, sondern vielmehr unter strikter Kontrolle stehen. So wird bei der Urease-Bildung das Nickel durch ein „Metallchaperon“ herangeschafft.
Harnstoff ist das Endprodukt des Stickstoff-Stoffwechsels bei Säugern (bei Vögeln: Harnsäure, bei Fischen: Ammoniak). Harnstoff tritt jedoch nicht nur als Abfallstoff auf, sondern er ist als weltweit wichtigster Dünger von großer volkswirtschaftlicher und ökologischer Bedeutung. Bei der technischen Synthese wird Ammoniak und Kohlendioxid bei erhöhter Temperatur miteinander umgesetzt. Urease katalysiert umgekehrt den Zerfall unter Freisetzung von Ammoniak. Die katalysierte Reaktion ist die Hydrolyse einer ersten C-N-Bindung. Das dabei entstehende Carbamat zerfällt anschließend in unkatalysierter Reaktion weiter zu Hydrogencarbonat und Ammoniak:
Während diese von der Urease verschiedener Bakterien katalysierte Reaktion die Ausscheidungen der Säuger wieder in den Stickstoff-Kreislauf überführt, ist die Wirkung auf den als Dünger hergestellten Harnstoff unerwünscht. Man schätzt, dass ca. die Hälfte des produzierten Düngers auf diese Weise verloren geht. Der entstehende Ammoniak ist als Stickstoffdünger nicht nur eingeschänkt verfügbar, er schädigt vielmehr die Wurzeln von Pflanzen – siehe hierzu auch die Wirkung des von Helicobacter-pylori-Urease erzeugten Ammoniaks im menschlichen Magen.
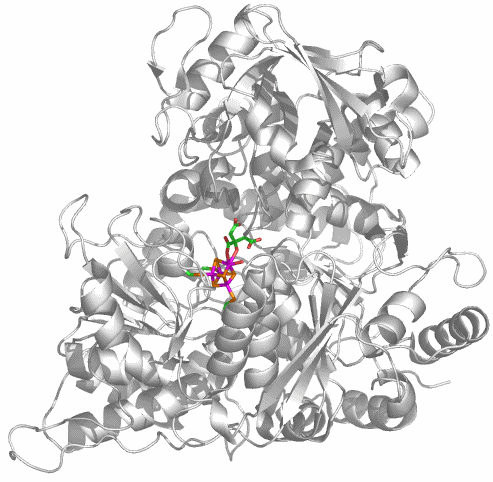
Urease besteht aus drei verschieden großen Untereinheiten. Bei der abgebildeten Klebsiella-aerogenes-Urease sind die Untereinheiten 556, 101 und 100 Aminosäuren lang. Es sind ca. 40 Strukturanalysen an Urease und Urease-Hemmstoff-Komplexen in der PDB hinterlegt. Proteindatenbank-Eintrag: 1EJX. Die Strukturanalyse liegt in einer Auflösung von 1.6 Å vor.
Holoenzym:

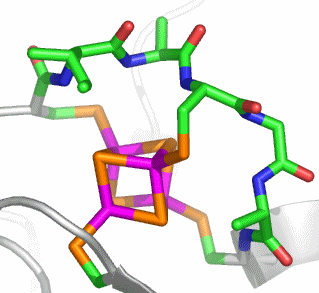
Bei der Betrachtung des aktiven Zentrums fällt ein carboxylierter Lysin-Rest auf, wodurch die CO2-Abhängigkeit von Urease verständlich wird:
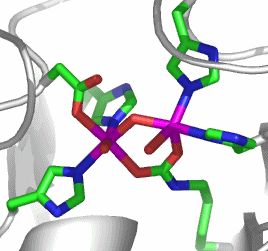
Wie bei der Hydrolyse polarer Bindungen durch hydrolytische Zink-Enzyme geht es auch bei der Urease darum, Hydroxid als Nukleophil zur Verfügung zu stellen – hier zum Angriff auf das Kohlenstoffatom einer polaren C-N-Bindung. Die Natur hat bei der Verwendung von Nickel anstelle von Zink zwei gleichsinnig wirkende Effekte auf die Lewis-Acidität des Metallzentrums auszugleichen: Nickel(II) ist weniger sauer als Zink(II), und es neigt aufgrund der Ligandenfeldstabilisierungsenergie (LFSE) von −12 Dq sehr viel deutlicher als Zink zur Ausbildung einer oktaedrischen Umgebung. Beides, die geringere Acididät und die Festlegung auf eine höhere Koordinationszahl, führt dazu, dass von einem einkernigen Nickelzentrum nicht die Stabilisierung eines Hydroxido-Liganden erwartet werden darf. Die Problemlösung der Natur ist einfach und wirksam: Urease enthält ein zweikerniges Metallzentrum, bei dem die Lewis-Aciditäten zweier Nickelatome zusammenwirken. Das für die C-N-Bindungsspaltung nötige OH−-Nukleophil liegt als Brückenligand vor, der nach Bindung des Substrats zu einem ortho-Diamidohydrogencarbonat-Übergangszustand bzw. einer entsprechenden Zwischenstufe führt. Deren anschließender Zerfall ergibt die Produkte Ammoniak und Carbamat.
Diamidophosphat (in manchen Publikationen „Phosphoramidat“ genannt), ist ein wirksamer Urease-Hemmstoff. Die tetraedrische Molekülstruktur entspricht weder der Struktur von Harnstoff noch von Carbamat, so dass geläufige Hemm-Mechanismen entfallen: so simulieren Hemmstoffe oft das Edukt oder das Produkt und weisen gleichzeitig eine höhere Bindungsenergie an das aktive Zentrum des Katalysators auf als diese. Es entsteht eine thermodynamische Falle, die Katalyse kommt zum Erliegen. Diamidophosphat (rechte Formel) hat dagegen Ähnlichkeit mit ortho-Diamidohydrogencarbonat (linke Formel), das als energiereiche Zwischenstufe oder als Übergangszustand formuliert worden war: Diamidophosphat ist ein Übergangszustands-Analog. Im Gegensatz zur Edukt- oder Produkt-Hemmung beruht die inhibierende Wirkung nicht nur auf einer festen Bindung im aktiven Zentrum, sondern darin, dass der nachfolgende Reaktionsschritt nicht zur Verfügung steht, um das aktive Zentrum wieder zu räumen (Diamidophosphat ist ein stabiles Molekül, das im Gegensatz zum regulären, instabilen Zwischenprodukt ortho-Diamidohydrogencarbonat nicht zerfällt).
Die Struktur eines Komplexes aus Diamidophosphat und Bacillus-Pasteurii-Urease ist im PDB-Eintrag 3UBP niedergelegt. Die Strukturanalyse liegt in einer Auflösung von 2.0 Å vor.
Hydratisierte Metall-Ionen sind viel zu reaktiv, als dass sie in der Zelle unkontrolliert herumschwimmen dürften. Übergangsmetalle wie Nickel, Eisen oder Kupfer unterliegen vielmehr strikter Kontrolle durch Transport- und Speicherproteine. Die Stabilitätskonstanten von Apoenzym-Metall-Komplexen und von Transportprotein-Metall-Komplexen sind in der Regel so groß, dass rechnerisch kein freies Metall-Ion in einer Zelle vorhanden ist. Der Mechanismus der Metallierung eines Apoproteins durch „sein“ passendes Transportprotein („Metallchaperon“) mit „seinem“ Metall ist derzeit ein intensiv untersuchter Vorgang. Die Nickelatome der Urease werden dem Apoenzym durch das nickelbindende Protein UreE eingepflanzt. Es liegt eine mäßig aufgelöste (ca. 3 Å) Strukturanalyse an nickelbeladenem UreE vor ([ur_uree_2010]). Das Apoprotein ist aus vier Proteinketten zusammengesetzt, die alle an der Nickelbindungsstelle beteiligt sind.
Struktur des Holoenzyms in 3NY0:
Das aktive Zentrum zeigt ein Nickel-Ion in einer gekappt oktaedrischen Umgebung von fünf His-Seitenketten. Wegen der mäßigen Auflösung darf wohl davon ausgegangen werden, dass ein sechster Ligand (Wasser?) vorhanden ist:
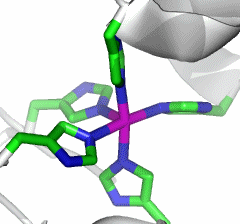
Wir überlegen, warum diese Ligandausstattung Nickel besser binden könnte als Eisen(II), Eisen(III), Kupfer(II) und Zink(II).
Urease war das erste Enzym, das kristallisiert wurde [ur_crystals_1926]. J. B. Sumner beschreibt oktaedrische Kristalle, die er aus Enzympräparaten der Jackbohne (“jack bean”, Canavalia ensiformis L.) gewinnt, und die im polarisierten Licht keine Doppelbrechung zeigen. Aus der chemischen Analyse leitet Sumner ab, dass Enzyme als einzige Bausteine Aminosäuren enthalten, also nicht etwa Kohlenhydrate oder eben Metalle. Dieser falsche Befund wurde erst ein halbes Jahrhundert später korrigiert [ur_nickel_1975] und [ur_nickel2_1975]. Erst 2010 gelang dann die Kristallstrukturbestimmung an Jackbohnenurease ([ur_jackbean_2010]).
Bei der Klausur … : Einschränkung der Koordinationsgeometrie durch Ligandfeldstabilisierungsenergie (LFSE); Darstellung einer Inhibitorwirkung im Energieschema einer Reaktion; Acidität und Nukleophilie verbrückender Aqua/Hydroxido-Liganden.
[ur_uree_2010]
R. Shi, C. Munger, A. Asinas, S. p. L. Benoit, E. Miller, A. Matte, R. J. Maier, M. Cygler:
Crystal Structures of Apo and Metal-Bound Forms of the UreE Protein from Helicobacter pylori: Role of Multiple Metal Binding Sites.
Biochemistry 2010, 49, 7080–7088.
doi:
10.1021/bi100372h
[ur_jackbean_2010]
A. Balasubramanian, K. Ponnuraj:
Crystal Structure of the First Plant Urease from Jack Bean: 83 Years of Journey from Its First Crystal to Molecular Structure.
J. Mol. Biol. 2010, 400, 274–283.
doi:
10.1016/j.jmb.2010.05.009
[ur_nickel_1975]
N. E. Dixon, C. Gazzola, J. J. Watters, R. L. Blakeley, B. Zerner:
Inhibition of jack bean urease (EC 3.5.1.5) by acetohydroxamic acid and by phosphoramidate. Equivalent weight for urease.
J. Am. Chem. Soc. 1975, 97, 4130-4131.
doi:
10.1021/ja00847a044
[ur_nickel2_1975]
N. E. Dixon, C. Gazzola, R. L. Blakeley, B. Zerner:
Jack bean urease (EC 3.5.1.5). Metalloenzyme. Simple biological role for nickel.
J. Am. Chem. Soc. 1975, 97, 4131-4133.
doi:
10.1021/ja00847a045
[ur_crystals_1926]
J. B. Sumner:
The Isolation and Crystallization of the Enzyme Urease.
J. Biol. Chem. 1926, 69, 435–441.
Die für die Beseitigung von Hyperoxid-Radikalen O2• − („Superoxid“) zuständigen Superoxid-Dismutasen MnSOD und FeSOD (es gibt außerdem die strukturell andersartigen CuZnSOD und NiSOD) zeigen mit einem einfachen, einkernigen Metallzentrum grundlegende Regeln der Redoxkatalyse: Am Metallzentrum müssen die passenden Oxidationsstufen zur Verfügung stehen; passend heißt, dass sie unter physiologischen Bedingungen die benötigten Redoxpotentiale aufweisen, und dass die Elektronenbilanz mit dem Redoxprozess am Substrat übereinstimmt.
Superoxid-Dismutasen sind wie die Katalasen Enzyme, deren Aufgabe die Beseitigung reaktiver Spezies darstellt, die den Organismus schädigen könnten. Superoxid entsteht beim Kontakt von Sauerstoff mit 1-Elektronen-Reduktionsmitteln wie den reduzierten Formen von Chinonen, Flavinen, Eisen-Schwefel-Clustern und Cytochromen vor allem in den Mitochondrien. Die gebildete Menge ist recht hoch, als typischer Wert werden 5 % der Elektronen der Atmungskette angegeben, die „fehlgeleitet“ werden. O2• − ist ein besonders reaktives Teilchen, das in reiner Form keine wässrige Lösungschemie aufweist – Superoxid disproportioniert sofort. So zerfällt oranges KO2 bei Wasserzugabe in stürmischer Reaktion unter Bildung von Sauerstoff und Peroxid. Im Enzym wird diese ohnehin rasche Reaktion um mehrere Größenordnungen beschleunigt, was die Brisanz des Superoxids unterstreicht. Unter Berücksichtigung der Aciditäten (pKA-Werte: HO2 4.8; H2O2 12) kann die katalysierte Reaktion für den physiologischen pH-Wert formuliert werden gemäß
2 O2•− + 2 H+ → O2 + H2O2
Mangan-Superoxid-Dismutase (MnSOD) scheint in der Evolution aus der älteren FeSOD hervorgegangen zu sein. Beiden Enzymen gemeinsam ist deren elektrochemisches Potential (MnSOD: 0.29 V; FeSOD: 0.22 V). Diese Gemeinsamkeit überrascht, da die Standardpotentiale der Redoxpaare Mn2+/3+ und Fe2+/3+ recht verschieden sind und man für eine derartige Anpassung eine deutlich verschiedene Ligandausstattung hätte erwarten dürfen. Angesichts des sehr weitgehend identischen Molekülbaus (siehe unten) überrascht eine Eigenart: Wird FeSOD von Eisen befreit und mit Mangan rekonstituiert oder wird MnSOD von Mangan befreit und mit Eisen rekonstituiert, so werden inaktive Enzyme erhalten.
Das Vorkommen der verschiedenen SOD-Typen ist bei CuZnSOD beschrieben.
Es liegen ca. 30 Strukturanalysen an MnSODs vor. Die hier gezeigte Escherichia-Coli-MnSOD ist ein Homotetramer aus vier Untereinheiten. Jeder einzelne Proteinstrang ist 205 Aminosäuren lang. Proteindatenbank-Eintrag: 1D5N. Die Auflösung beträgt 1.55 Å, es ist eine der vier Untereinheiten gezeigt.
Holoenzym:
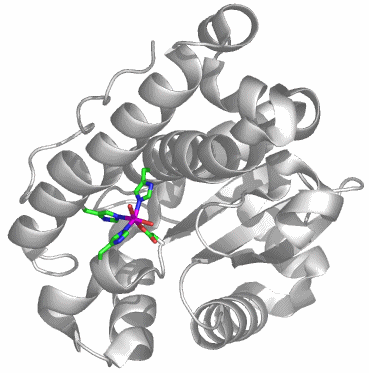
Im aktiven Zentrum ist ein Mangan(III)-Zentralatom von drei Histidin-, einem Aspartat- und zwei Aqua/Hydroxido-Liganden koordiniert:
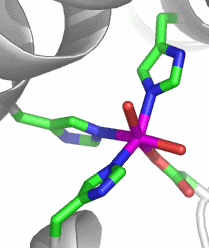
Im ersten Schritt des Katalysecyclus bindet ein Superoxid-Ion an das Manganzentrum und erhöht dabei dessen Koordinationszahl auf sechs [mnsod_2010]. Anschließend kommt es zur Elektronenübertragung und Ligandprotonierung. Es entsteht Mangan(II) und Sauerstoff, der das Metallzentrum verlässt.
Wie bei jeder Redox-Katalyse wird natürlich auch bei den SODs die thermodynamische Randbedingung eingehalten, dass das elektrochemische Potential des aktiven Zentrums zwischen den Potentialen der beiden Halbreaktionen des katalysierten Prozesses liegen muss:
Das Ergebnis, dass die Potentiale bei MnSOD und FeSOD nahezu gleich sind, ist sehr überraschend, da (1) die Standardpotentiale weit auseinanderliegen (FeII/III 0.77, MnII/III 1.41 V bei pH=0) und (2) die Umgebung der Metallzentren in beiden Enzymen praktisch dieselbe ist, so dass der Abstand der Potentiale von ungefähr einem halben Volt als gewahrt zu erwarten gewesen wäre. Offensichtlich aber sind die aktiven Zentren weniger ähnlich als die Betrachtung der Strukturanalysen erwarten lässt. Wird nämlich MnSOD von Mangan befreit und mit Eisen rekonstituiert, so sinkt das Potential um ca. ½ Volt, und umgekehrt wird in mangansubstituierter FeSOD ein um mehr als ½ Volt erhöhtes Potential gemessen. Beide mit dem „falschen“ Metall rekonstituierte SODs sind aufgrund dieser Potentialverschiebungen inaktiv. Die MIII-Form von Fe-substituierter MnSOD hat ein zu kleines Potential (ist zu schwach oxidierend), um Superoxid zu Sauerstoff zu oxidieren, während die MII-Form Mn-substituierter FeSOD ein zu hohes Potential hat (zu schwach reduzierend wirkt), um Superoxid zu H2O2 reduzieren zu können. Die strukturelle Ursache ist Gegenstand der aktuellen Diskussion. Es scheint wahrscheinlich, dass geringfügige Unterschiede in der zweiten Koordinationsspäre der aktiven Zentren ursächlich sind. [mnsod1]
Wir betrachten den Katalysecyclus und vor allem die Metall(III)-Ruheform, bei der wir uns fragen, warum nicht die ebenfalls im Zyklus vertretene Metall(II)-Stufe als Ruheform vorliegt – vor allem bei Mangan. Hierzu schauen wir auf die Komplexbeständigkeit und unterscheiden metallabhängige Enzyme von Metalloenzymen.
Bei der Klausur … : Faustregeln kennen, um die Stabilität von Komplexen abzuschätzen; LFSE-Werte ausrechnen; Jahn-Teller-Verzerrung; Umgang mit elektrochemischen Potentialen.
[mnsod_2010]
J. Porta, A. Vahedi-Faridi, G. E. O. Borgstahl:
Structural Analysis of Peroxide-Soaked MnSOD Crystals Reveals Side-On Binding of Peroxide to Active-Site Manganese.
J. Mol. Biol. 2010, 399, 377–384.
doi:
10.1016/j.jmb.2010.04.031
Viele Regeln der Redox-Katalyse lassen sich an zwei besonders einfachen aktiven Zentren erkennen, in denen ein einzelnes Eisen-Zentralatom zwischen den geläufigen Oxidationsstufen +II und +III wechselt: das Fe(SCys)4-Zentrum in Rubredoxin (Rd) und das Fe(NHis)4(SCys)-Zentrum in Superoxid-Reduktasen (SOR). Da beide Zentren in allen Oxidationszuständen in der high-spin-Form vorliegen, lässt sich der Einfluss der Liganden auf die elektrochemischen Potentiale allein untersuchen.
Während Rubredoxin (Rd) strukturell zu den am besten untersuchten Metalloenzymen gehört, lag seine Funktion lange im Dunklen. In den letzten Jahren verdichten sich die Hinweise, dass Rd bei der Superoxid-Beseitigung in Anaerobiern beteiligt ist. Anaerobiern nützt eine SOD nur bedingt, da für diese Organismen auch Sauerstoff giftig ist. An die Stelle der SODs treten hier SORs, Superoxid-Reduktasen. Superoxid wird durch diese zu H2O2 reduziert, als finales Reduktionsmittel dient NAD(P)H:
O2•− + 2 H+ + e− → H2O2
Für die Elektronenübertragung scheinen drei Katalysatoren verantwortlich zu sein. Die eigentlichen SORs übertragen 1 Elektron auf Superoxid; die oxidierte Form SOR(ox) wird anschließend durch Rd wieder reduziert, Rd(ox) wird anschließend durch die Vermittlung einer NADPH:Rubredoxin-Oxidoreductase reduziert, diese wiederum erhält das Elektron von NAD(P)H (Schema nach [sor_2005]:
Superoxidreduktasen („Neelaredoxin“ und andere) werden durch Rd reduziert [sor_2005]. Neelaredoxin ist eine SOR aus Pyrococcus furiosus. P. furiosus ist ein Archeon, das in ca. 100 °C heißer Umgebung lebt – so wie am Entdeckungsort, der italienischen Insel Vulcano nördlich von Sicilien. Hinter der Untersuchung von Hyperthermophilen-Enzymen steht nicht nur wissenschaftliches Interesse, auch technisch versprechen diese extrem temperaturbeständigen Proteine ein breites Anwendungsfeld.
Während für Neelaredoxin neben der SOR- auch SOD-Aktivität beschrieben wird, scheint ein verwandtes Enzym, Desulfoferroredoxin (Dfx) allein SOR-aktiv zu sein.
Clostridium-pasteurianum-Rubredoxin in der Eisen(III)-Form in 1.5 Å Auflösung (PDB-Eintrag: 1FHH, zum Vergleich die Eisen(II)-Form): 1FHM):
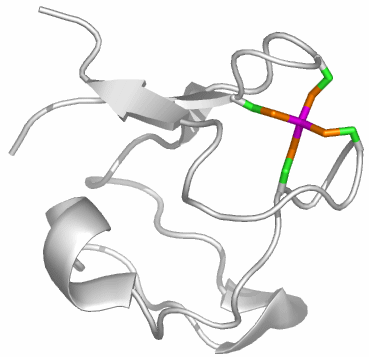
Rubredoxin ist ein sehr kleines Protein. Das dargestellte C.-pasteurianum-Enzym ist aus nur 54 Aminosäuren aufgebaut. Vier deprotonierte Cystein-Reste koordinieren ein Eisen(III)-Zentrum in einem mittleren Fe-S-Abstand von 2.27 Å. In der reduzierten Form wird ein Mittelwert von 2.36 Å gefunden. Die Differenz von 0.09 Å ist im Vergleich mit Modellverbindungen eher groß. Ein vergleichbares Paar von Pyrococcus-furiosus-Rubredoxin-Strukturen ergibt Mittelwerte von 2.27 und 2.32 Å. Die Differenz von 0.05 Å entspricht hier derjenigen in niedermolekularen Thiolatoferraten; sie ist deutlich geringer als die Differenz der Ionenradien für tetraedrisch koordiniertes Eisen(II/III) von 0.14 Å.
Die Struktur von Rd enthält ein konservatives Bauelement. Sowohl bei Rd als auch bei vielen anderen Eisen-Schwefel-Proteinen werden die vier Cysteinat-Liganden meist von zwei Cys-X-X-Cys-Motiven bereitgestellt. Für X können beliebige Aminosäuren auftreten, charakteristisch ist jedoch die Zahl der verbrückenden Aminosäuren, die der Klassifizierung von Rubredoxinen dient: Bei Typ-I-Rds liegen immer zwei Aminosäuren in der Brücke vor, bei Typ-II-Rds gibt es neben einem Cys-X-X-Cys- auch ein Cys-X-X-X-X-Cys-Motiv. In C.-pasteurianum-Rd ergibt sich das folgende Bild (vom Rest des Enzyms ist dünn nur die Proteinhauptkette dargestellt):
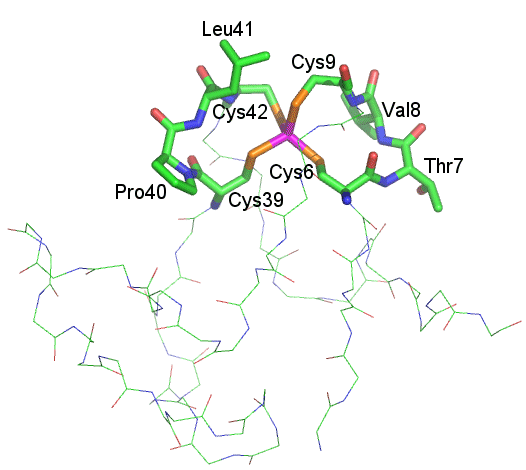
Das Potential von Rd beträgt ca. 0 V. Es entspricht damit ungefähr sowohl dem FeII/III-Potential in wässriger Lösung als auch dem Potential vergleichbarer löslicher Komplexe wie zum Beispiel [FeII/III(ox)3]4/3−. Alle diese Werte liegen erwartungsgemäß deutlich unter dem Standardpotential im Sauren von +0.77 V. Die Steuerung eines Potentials durch die Ladung der Liganden lässt sich bei Rd beispielhaft darstellen. [sor_1998] stellt in Figure 9 das Potential von Rd-Mutanten über den möglichen pH-Bereich dar, in dem die Enzyme stabil sind. Die einzelnen Mutanten sind Cys6Ser, Cys9Ser und Cys42Ser, es wurde also jeweils eine eisenbindende Cystein-Position gegen Serin ausgetauscht:
Als Ergebnis wird ein pH-unabhängiges Potential für Wildtyp-Rd beobachtet, während die Potentiale der Mutanten bei niedrigem pH-Wert mit ca. 0.06 V pro pH-Einheit abfallen, bis sie zur Konstanz gelangen. Der Zahlenwert von 0.06 V pro pH-Einheit entspricht gemäß der Nernstschen Gleichung einer Kopplung einer 1-Protonen-Übertragung an die Übertragung von 1 Elektron. Dieselbe Abhängigkeit zeigt zum Beispiel das wässrige FeII/III-System im alkalischen Bereich. Berücksichtigt man die pKA-Werte der beteiligten Spezies, indem man die Absenkung der pKA-Werte der freien Liganden (Cys 8.3, Ser ca. 15) bei der Bindung an die beiden unterschiedlich Lewis-sauren Zentren Eisen(II) und Eisen(III) berücksichtigt, so wird über weite Bereiche aus dem pH-unabhängigen Gleichgewicht
[FeII(SCys)4]2− ⇄ [FeIII(SCys)4]− + e−
das pH-abhängige Gleichgewicht
[FeII(HOSer)(SCys)3]− ⇄ [FeIII(OSer)(SCys)3]− + e− + H+
Es wird eine allgemeine Regel sichtbar, welche die Natur oft zur Abstimmung von elektrochemischen Potentialen nutzt: anionische Liganden stabilisieren die höhere Oxidationsstufe des Redoxpaars. Stabilisierung bedeutet Erniedrigung der Oxidationskraft der höheren Oxidationsstufe, dies entspricht einem Absinken des elektrochemischen Potentials.
Der Oxidationszustand von Rd lässt sich leicht durch UV/Vis-Spektroskopie verfolgen. Rd-Spektren sind zum Beispiel in [sor_2005] abgebildet (Fig. 2; man beachte bei der Abschätzung der Farbintensität die Konzentrationsangabe mm statt des üblichen m bei der Ordinateneinteilung). Die starke Absorption der oxidierten Form im Grünen, die zur roten Farbe des Enzyms führt (Rubredoxin von lat. rubrum, rot) ist ein LMCT-Übergang von einem Thiolato-Ligand auf Eisen(III). Das Fehlen von Kristallfeldübergängen wird ebenso diskutiert wie der Spinzustand der Eisenzentren.
Es liegen Strukturanalysen von Pyrococcus-furiosus-Neelaredoxin in der oxidierten und in der reduzierten Form bei 2 Å Auflösung vor. Die reduzierte Form ist in 1DQK beschrieben. Von den vier gleichartigen, je 124 Aminosäuren langen Proteinsträngen in der asymmetrischen Einheit ist ein Strang dargestellt:
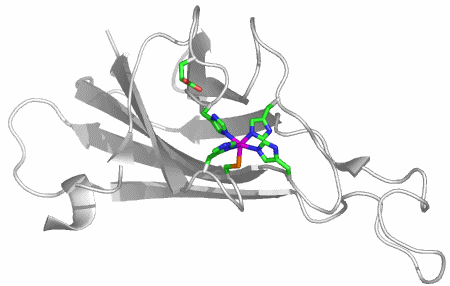
Das aktive Zentrum von Nlr in der reduzierten Form ist zusammen mit einem entfernten Glutamat und einem Lysinrest dargestellt. Der Glutamat-Rest ist in der oxidierten Form an Eisen gebunden, auf die mögliche Bedeutung des Lysinrestes wird beim Katalysecyclus eingegangen:
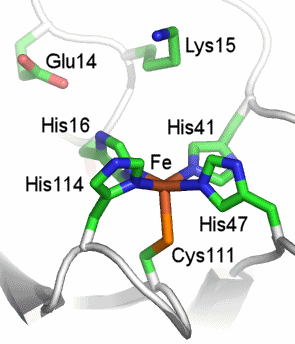
Die oxidierte Form ist in 1DO6 niedergelegt. Bei zwei der vier Proteinstränge der asymmetrischen Einheit ist das Eisen(III)-Atom im aktiven Zentrum sechsfach koordiniert:
Eine neuere Publikation [sor_2007] zeigt die Peroxid-beladene SOR von Mutanten von Desulfoarculus baarsii bei einer Auflösung von 1.95 Å (2JI3). Raman-Spektroskopie am oxidierten und Wasserstoffperoxid-getränkten Kristall zeigen ein (FeIII-OOH)-Zentrum:
Das Fe(His)4Cys-Zentrum wird – auch in Anwesenheit von Sauerstoff – bei Nlr und Dfx üblicherweise in der Eisen(II)-Form isoliert. Im Einklang hiermit ist das elektrochemische Potential mit 0.2–0.3 V zu hoch, als dass Sauerstoff in einem 1-e-Schritt, also unter Reduktion zu Superoxid, ein SOR-Zentrum in die dreiwertige Stufe oxidieren könnte. Das SOR-Potential entspricht denjenigen von MnSOD und FeSOD. In Übereinstimmung hiermit steht die (allerdings umstrittene, siehe unten) SOD-Aktivität von Nlr. Nlr wird aufgrund der Potentiallage erst durch die Anwesenheit von Rd, von dem es 1 Elektron aufnehmen kann, zur SOR. (Im folgenden Schema bezieht sich die Nummerierung von Aminosäuren auf Nlr, nicht Dfx.)
Bei Anwesenheit von Rd startet der Katalysecyclus mit der Eisen(II)-Form. Superoxid bindet an die freie Koordinationsstelle des quadratisch-pyramidal koordinierten Zentralatoms. Dieses wird zur dreiwertigen Stufe oxidiert, der Ligand ist nun eine Hydroperoxido-Gruppe. Unterstützt durch die Bindung eines entfernten Glutamats wird H2O2 abgespalten, und das Enzymzentrum geht in die isolierbare, dreiwertige Ruheform über. Ist Rd vorhanden, findet im nächsten Schritt Reduktion in die zweiwertige Ruheform statt. Ist der nächste Reaktand ein weiteres Superoxid-Ion, so reduziert dieses an der Stelle von Rdred das Eisenzentrum. Anstatt Rdox verlässt bei diesem SOD-Weg O2 den Cyclus. Die thermodynamisch mögliche SOD-Aktivität von Neelaredoxin ist in der Literatur nicht unumstritten. Möglicherweise ändern weitere Untersuchungen die Aussagen zum SOD-Zweig des Katalysecyclus.
Der Cyclus zeigt, dass für eine erfolgreiche Katalyse ein Proton-Shuttle zur Verfügung stehen muss. Die derzeitige Vorstellung ist die, dass eines der beiden benötigten Protonen über das nahe liegende Lys15 transportiert wird, während das zweite Proton direkt aus der Umgebung stammt. Bei dieser Einschätzung wird die Exposition des aktiven Zentrums berücksichtigt, das nahe an der Proteinoberfläche liegt. Ein zu Beginn protoniertes Lys15 dient als kationische Position der schnellen Weiterleitung der Superoxid-Anionen in das aktive Zentrum.
Der Spinzustand der Eisenatome scheint während des gesamten Cyclus – auch in der Peroxido-Form – high-spin zu sein. Die Aussage ist nicht völlig sicher, da Rechnungen an einer end-on-gebundenen Hydroperoxido-Form auch den low-spin-Fall zulassen. Bis das Gegenteil wahrscheinlicher ist, diskutieren wir die Elektrochemie des Nlr-Zentrums nur für die high-spin-Situation. Diese ist für Rd aufgrund der tetraedrischen Koordination des Fe(SCys)4-Zentrums und der Stellung von Thiolat in der spektrochemischen Reihe sicher. Bei der Beurteilung des Rd-Zentrums als katalytisch aktivem Strukturelement ist die high-spin-Situation kritisch zu sehen. Die Differenz der high-spin-Ionenradien von FeII und FeIII ist nämlich mit 0.14 Å recht groß. Damit ist auch eine hohe Reorganisationsenergie zu erwarten, die im Redoxprozess als Aktivierungsenergie erscheint. Eine nennenswerte Aktivierungsschwelle widerspricht aber der Idee einer wirksamen Katalyse. Im Einklang mit dieser Feststellung wurden bislang keine elektronenleitenden Zentren beschrieben, die sich auf weitgehend ionische high-spin-Eisen(II/III)-Zentren stützen. Im Fall des Rubredoxins ist die Kovalenz der Eisen-Schwefel-Bindungen zu beachten, die zu einer hinreichend geringen Bindungslängenänderung führt.
Das ungewöhnliche quadratisch-pyramidale FeN4S-Zentrum von Nlr wurde in einem weiteren Enzym gefunden, dem Desulfoferrodoxin (Dfx). Eine Strukturanalyse an Desulfovibrio-desulfuricans-Dfx (1DFX) zeigt eine 125 Aminosäuren lange Proteinkette und 2 einkernige Eisenzentren: ein Zentrum I, das einem Rubredoxin-Zentrum entspricht, und ein Zentrum II, das dem aktiven Zentrum von Nlr gleicht. Da Unsicherheit über die Funktion von zwei Eisenzentren herrscht, gehen wir in dieser Vorlesung vorerst nicht näher auf Dfx ein.
Bei der Klausur … : Einfluss von Liganden auf elektrochemische Potentiale, Bedeutung von Potentiallagen in Reaktionskaskaden, Reorganisationsenergie.
[sor_2007]
G. Katona, P. Carpentier, V. Nivière, P. Amara, V. Adam, J. Ohana, N. Tsanov, D. Bourgeois:
Raman-Assisted Crystallography Reveals End-On Peroxide Intermediates in a Nonheme Iron Enzyme.
Science 2007, 316, 449–453.
doi:
10.1126/science.1138885
[sor_2005]
J. V. Rodrigues, I. A. Abreu, L. M. Saraiva, M. Teixeira:
Rubredoxin acts as an electron donor for neelaredoxin in Archaeoglobus fulgidus.
Biochem. Biophys. Res. Commun. 2005, 329 1300–1305.
doi:
10.1016/j.bbrc.2005.02.114
[sor_1998]
Z. Xiao, M. J. Lavery, M. Ayhan, S. D. B. Scrofani, M. C. J. Wilce, J. M. Guss, P. A. Tregloan, G. N. George, A. G. Wedd:
The Rubredoxin from Clostridium pasteurianum: Mutation of the Iron Cysteinyl Ligands to Serine. Crystal and Molecular Structures of Oxidized and Dithionite-Treated Forms of the Cys42Ser Mutant.
J. Am. Chem. Soc. 1998, 120, 4135–4150.
doi:
10.1021/ja973162c
[2Fe-2S]-Ferredoxine und Rieske-Zentren, beides verbreitete 1-Elektronen-Überträger, sind sowohl strukturell als auch hinsichtlich der Oxidationsstufen nah verwandt – Rieske-Zentren übertragen jedoch bei deutlich höherem Potential 1 Elektron. Die Bedeutung der Ligandladung zur Steuerung des elektrochemsichen Potentials lässt sich an diesem Enzympaar besonders deutlich herausarbeiten.
[2Fe-2S]-Ferredoxine ([2Fe-2S]-Fds) sind ubiquitäre 1-Elektron-Überträger bei niedrigem elektrochemischen Potential (ca. −0.4 bis −0.1 V). So übernehmen [2Fe-2S]-Fds in grünen Pflanzen als lösliche Elektronenüberträger in den Chloroplasten Elektronen vom Photosystem I. Auch Rieske-Zentren, die als Enzyme der Atmungskette und der Photosynthese ebenfalls ubiquitär sind, geben jeweils 1 Elektron weiter, jedoch bei einem höheren Potential von ca. +0.3 V.
Oxidierte Form von [2Fe-2S]-Ferredoxin aus Spinacia oleracea (PDB-Eintrag: 1A70), Auflösung: 1.7 Å:
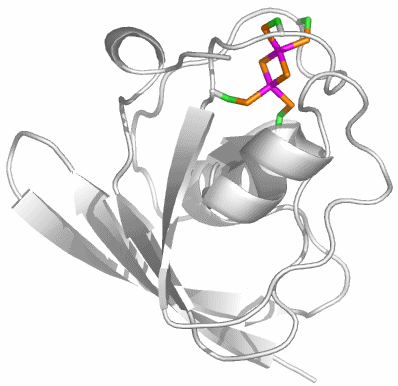
Das aktive Zentrum besteht aus einer Fe2S2-Raute, deren Eisenatome durch jeweils zwei Cysteinato-Liganden tetraedrische Koordination erreichen.
Gezeigt ist die Struktur der löslichen Domäne des Rieske-Proteins II aus Sulfolobus acidocaldarius in der Eisen(III)-Form (PDB-Eintrag: 1JM1), Auflösung: 1.1 Å:
Das aktive Zentrum moduliert das Bauprinzip der [2Fe-2S]-Fds, indem zwei Cysteinato-Liganden in asymmetrischer Weise durch zwei Histidin-Liganden ersetzt sind:
Mittlere Atomabstände: im FeS4-Fragment: Fe-SCys 2.34, Fe-SSulfid 2.26; im FeS2N2-Fragment: Fe-SSulfid 2.26, Fe-N 2.09 Å.
Beide Zentren reagieren auf demselben Oxidationsstufen-Niveau. [2Fe-2S]-Fds katalysieren den Fluss eines Elektrons gemäß:
Cys2FeIIIS2FeIICys2 ⇄ Cys2FeIIIS2FeIIICys2 + e−
Rieske-Zentren verhalten sich gleich, wobei das redoxaktive Zentrum das FeN2S2-Tetraeder ist:
Cys2FeIIIS2FeIIHis2 ⇄ Cys2FeIIIS2FeIIIHis2 + e−
Aus den im vorigen Kapitel abgeleiteten Gesetzmäßigkeiten ergibt sich, dass sich in der FeIIIFeII-Form das Eisen(III)-Atom im S4-Tetraeder befindet.
Fe2S2-Cluster, vor allem aber Fe4S4-Cluster, lassen sich ohne sonderlichen Aufwand synthetisieren. Neben der eigentlichen Motivation zur Synthese von Modellverbindungen wie der genaueren Kenntnis struktureller, spektroskopischer oder magnetischer Einzelheiten, wirft dies Licht auf Hypothesen zur Beteiligung von Eisensulfiden wie FeS oder FeS2 bei der Entstehung des Lebens. Neben das Prinzip der genetischen Festlegung tritt die Selbstorganisation funktionaler Baueinheiten, für die das Protein nur den äußeren Rahmen schafft.
Im Einklang mit der in-vitro-Synthese von Eisen-Schwefel-Clustern steht der Befund, dass Apo-[2Fe-2S]-Fds durch Eisen(II) und Sulfid wieder zu Holo-[2Fe-2S]-Fds rekonstituiert werden können. Diese Beobachtung gilt auch für die im nächsten Kapitel behandelten häufigeren [4Fe-4S]-Fds. Vor diesem Hintergrund überrascht die komplexe Biosynthese von Ferredoxinen. Die umfangreichste Information liegt für [2Fe-2S]-Zentren vor. Deren Synthese scheint in Säuger-Mitochondrien mit einem Eisen(II)-transportierenden Protein Frataxin (Ftx) zu beginnen, das Eisenatome auf einem Trägerprotein ISU (IS für Iron Sulfur, U zur Unterscheidung von ISU von anderen Proteinen des IS-Operons) ablegt. Durch zu Persulfiden aufgebaute Cysteine wird den Eisenatomen Sulfid zugefügt; die Ladung des Fe2S2-Clusters wird durch ein Fd eingestellt. Schließlich wird der intakte Fe2S2-Cluster auf Apo-Fd übertragen. Die Vorstellungen zum Holo-Fd-Aufbau sind zur Zeit nicht durch Kristallstrukturanalysen eines IS-Proteins oder von Fe-beladenem Ftx gestützt. Die auf den ersten Blick mit der aufwendigen Fd-Biossynthese kaum vereinbare Hypothese, dass archaische Eisen-Schwefel-Cluster durch Selbstorganisation entstanden sein könnten, gewinnt an Plausibilität, wenn Anaerobier und deren höhere Eisen(II)-Toleranz mit aerobem Lebem verglichen werden. Erst die Anwesenheit des in der Uratmosphäre nicht vorhandenen Sauerstoffs lässt Eisen(II) brisant werden (Superoxid-Bildung, Fenton-Reaktionen).
Bei der Klausur … : Kristallfeldaufspaltung im Tetraederfall, Auswahlregeln in Elektronenspektren, charge-transfer-Übergänge, pH-Abhängigkeit elektrochemischer Potentiale, Stabilisierung hoher/niedriger Oxidationsstufen.
Verwendete Literatur: siehe Lehreinheit „[4Fe,4S]-Ferredoxine und Aconitase“.
Die Delokalisiation der Oxidationszustände in [4Fe-4S]-Redoxproteinen geht mit kaum messbaren Änderungen der Struktur der Cluster im reduzierten und oxidierten Zustand einher. Die Reorganisationsenergie ist entsprechend klein und die Katalyse wirksam. Auch Fe4S4-Zentren werden in heutigen Organismen durch Proteine biosynthetisiert. Zugleich fällt bei Modellierungsstudien ihre hohe Bildungstendenz durch Selbstorganisation auf: der an vier Thiolate gebundene FeII2FeIII2S4-Kern der oxidierten Form eines Ferredoxins entsteht bereitwillig sowohl aus den Komponenten, aber auch bei Versuchen, den zweikernigen [(RS)2FeIIS2FeIII(SR)2]3−-Cluster zu synthetisieren unter Thiolat-Freisetzung. Vor dem Hintergrund dieser hohen Bildungstendenz wird über die Bedeutung von Eisen-Schwefel-Clustern bei der Entstehung des Lebens in der reduzierenden Uratmosphäre spekuliert. Dazu gehört auch die Nutzung dieses früh verfügbaren Zentrums für unterschiedliche Aufgaben. So erledigt noch heute ein vierkerniger Eisen-Schwefel-Cluster nach geringfügiger Anpassung des Baumotivs eine typische „Zink-Aufgabe“: Aconitase, ein metallabhängiges Metalloenzym.
[4Fe-4S]-Cluster sind Ferredoxin oder HP-Protein. [4Fe-4S]-Fds sind die ubiquitären 1-Elektronen-Schaltstellen in Mitochondrien und Chloroplasten bei niedrigem elektrochemischen Potential. Sie treten nach dem folgenden Schema entweder zwischen den Valenzzuständen Fdred/Fdox oder HPred/HPox auf, bisher wurden jedoch noch keine [4Fe-4S]-Cluster gefunden, die als 2-Elektronen-Überträger beide Stufen umfassen.
Als „Ferredoxine“ werden üblicherweise nur Proteine beschrieben, in denen das Redoxpaar bei niedrigem Potential realisiert ist. Das Redoxpaar bei hohem Potential bildet die Stufe der HP-Proteine, der High Potential Iron Proteins, in manchen Publikationen auch „HiPIPs“ abgekürzt.
Im Gegensatz zu den [2Fe-2S]-Fds liegen in [4Fe-4S]-Proteinen keine lokalisierten Valenzzustände vor. Im Einklang hiermit ist die Reorganisationsenergie von [4Fe-4S]-Clustern kleiner als die der dinuklearen Zentren [fd_redox_2004].
Dargestellt ist eine hochaufgelöste (1 Å) Strukturanalyse an der oxidierten [4Fe-4S]2+-Form von Bacillus-thermoproteolyticus-Fd (PDB-Code: 1IRO; ein HP-Protein ist zum Beispiel in 1IUA hinterlegt. Die Proteinstränge sind mit 80 (1IRO) bzw. 82 Aminosäuren (1IUA) sehr kurz:
Das aktive Zentrum zeigt die starke Abweichung von einem idealisierten Würfel. Der mittlere Fe-S-Abstand beträgt 2.27 Å. Wie schon Typ-I-Rubredoxine enthalten auch [4Fe-4S]-Ferredoxine durch zwei Aminosäuren getrennte Cysteine. So enthält 1IRO ein Cys-XX-Cys-XX-Cys-Muster:
Aconitase ist ein Enzym des Citrat-Cyclus. Sie katalysiert die Isomerisierung von Citrat zu Isocitrat durch Dehydratisierung zu cis-Aconitat und anschließender Re-Hydratisierung (Formeln siehe bei „Katalysecyclus“).
Bei der Aconitase ist der seltene Fall realisiert, dass fast alle Schritte des Katalysecyclus durch Strukturanalysen gefasst sind. Das Holoenzym ist erheblich komplexer als ein Ferredoxin. Abgebildet ist die Ser642Ala-Mutante von Rindermitochondrien-Aconitase, deren Proteinkette 753 Aminosäuren lang ist. (1.8 Å Auflösung, PDB-Eintrag: 1C96):
Der Fe3S4-Cluster im unvollständigen, inaktiven Zentrum von nativer Schweinemitochondrien-Aconitase (2.1 Å Auflösung, PDB-Eintrag: 5ACN):
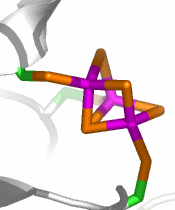
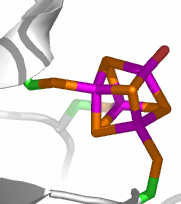
Der Eisen-Schwefel-Cluster nach der Rekonstitution mit Eisen(II); an das locker gebundene Eisen koordiniert ein Aqua-Ligand (2.1 Å Auflösung, PDB-Eintrag: 6ACN):
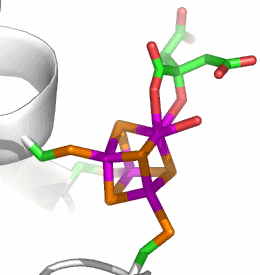
Ein Substrat-Enzym-Komplex konnte bei der oben als Holoenzym dargestellten, funktionell stark eingeschränkten Ser642Ala-Mutante von Rindermitochondrien-Aconitase kristallisiert werden. Citrat bindet als Chelatligand mit einer Carboxylat- und der alkoholischen Hydroxy-Funktion:
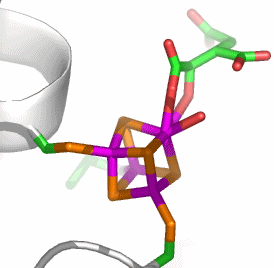
Dieselbe Ser642Ala-Mutante von Rindermitochondrien-Aconitase mit gebundenem Isocitrat (2 Å Auflösung, PDB-Eintrag: 1C97):
Der Katalysecyclus beginnt nach der Citrat-Anlagerung mit der Deprotonierung am Kohlenstoffgerüst durch eine Base, die meist als Serinat-642 angesehen wird. Das anschließend gebildete cis-Aconitat verschiebt seine Metallbindungsstelle, was ungefähr einer 180°-Drehung des Substrats im Enzymzentrum entspricht. Die Re-Hydratisierung führt zum Produkt Isocitrat.
Bei der Klausur … : Selbstorganisation, Mustererkennung, Spinkopplung in Clustern mit Brückenliganden, Superaustausch.
[fd_2004]
P. Venkateswara Rao, R. H. Holm:
Synthetic Analogues of the Active Sites of Iron–Sulfur Proteins.
Chem. Rev. 2004, 104, 527–560.
doi:
10.1021/cr020615+
[fd_biosynth_2004]
S. S. Mansy, J. A. Cowan:
Iron–Sulfur Cluster Biosynthesis: Toward an Understanding of Cellular Machinery and Molecular Mechanism.
Acc. Chem. Res. 2004, 37, 719–725.
doi:
10.1021/ar0301781
[fd_redox_2004]
M.-L. Tan, E. A. Dolan, T. Ichiye:
Understanding Intramolecular Electron Transfer in Ferredoxin:? A Molecular Dynamics Study.
The Journal of Physical Chemistry B 2004, 108, 20435–20441.
doi:
10.1021/jp046367y
Das Prinzip "ein Strukturelement – viele Funktionen" ist in der Natur verbreitet. Wir vergleichen hierzu Oxidodieisen-Zentren in löslicher Methan-Monooxygenase und im Sauerstoff-Transporter Hämerythrin (Hr). Methanotrope Bakterien, die Methan als einzige Kohlenstoffquelle benutzen können, verfügen mit der Hydroxylase-Komponente (sMMOH) des Enzymkomplexes in der löslichen Methan-Monooxygenase (sMMO) über Oxidodieisen-Zentren, an denen Methan durch Sauerstoff zu Methanol oxygeniert wird. Es ist verblüffend, dass eine nicht-triviale Reaktion wie die C-H-Aktivierung durch ein unspektakulär ausgestattetes zweikerniges Eisenzentrum katalysiert wird. Spektakulär ist hingegen die Beobachtung, dass im Katalysecyclus offensichtlich die Oxidationstufe +IV an den beiden Eisenatomen durchlaufen wird. Trotz der strukturellen Verwandtschaft erfüllt das sauerstoff-transportierende Oxidodieisen-Zentrum in Hr eine völlig unterschiedliche Aufgabe. Voraussetzung für die Enzymfunktion ist, dass ein höheres Redoxpotential realisiert ist als es dem Standardpotential des Eisen(II/III)-Halbelements bei pH 7 entspricht, dass zugleich aber die vierwertige Stufe nicht stabilisiert sein sollte.
Hr ist der Sauerstofftransporter wirbelloser Meerestiere.
Hr ist sowohl im unbeladenen als auch im O2-beladenen Zustand kristallographisch charakterisiert. Hr ist ein kleines Protein mit 113 Aminosäuren, die in charakteristischer Weise einen Stapel aus vier α-Helices bilden. Gezeigt ist dies am Beispiel des Hämerythrins von Themiste dyscritum, eines im Meer lebenden Wurms, das im beladenen (PDB-Code 1HMO) und unbeladenen Zustand (PDB-Code 1HMD) mit einer Auflösung von jeweils 2 Å analysiert wurde. Abgebildet sind zwei Ansichten des Säulenstapels der unbeladenen Form:
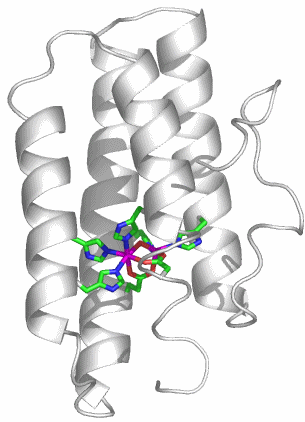
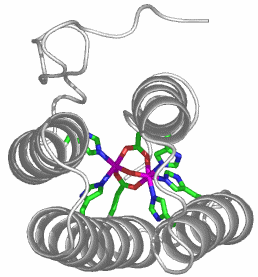
Das aktive Zentrum weist eine freie Koordinationsstelle auf; die Ligandausstattung besteht aus fünf His-Seitenketten, einem verbrückenden Hydroxido-Ligand, 1 Asp und 1 Glu:
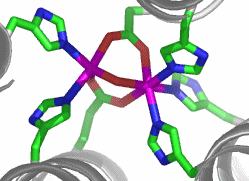
An diese Position bindet im beladenen Zustand das O2-Molekül
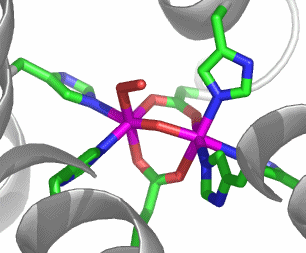
Quantenchemische Rechnungen zeigen, dass die Sauerstoff-Anlagerung ein Redox-Prozess ist, der durch eine Protonenverschiebung begleitet wird: Durch den Übertrag von zwei Elektronen entstehen zwei Eisen(III)-Zentren und ein Peroxid-Ion. Das Anion nimmt das Proton des verbrückenden Hydroxido-Liganden auf und betätigt eine Wasserstoffbrückenbindung zum gebildeten Oxido-Ligand. Bei der Freisetzung verlaufen die umgekehrten Vorgänge, Eisen(III) muss Peroxid zu Sauerstoff oxidieren können. Das Standardpotential in saurer Lösung für die Peroxid-Oxidation beträgt 0.7 V, bei pH 7 ergibt sich 0.3 V. Ohne Liganden beträgt das Eisen(II,III)-Potential bei pH 7 ca. 0 V, eine Sauerstoff-Freisetzung wäre unter diesen Bedingungen nicht möglich. Hr ist ein weiteres Beispiel, wie eine größere Zahl von His-Liganden das elektrochemische Potential so weit erhöht, dass die benötigte Oxidationskraft der oxidierten Form zur Verfügung steht.
Bei der magnetischen Untersuchung beider Hr-Formen fällt die antiferromagnetische Kopplung der spintragenden Eisenzentren auf (J = −13 und −77 cm−1 für deoxy- und oxy-Hr), die aufgrund der Strukturparameter auch erwartet werden darf [hr_2004].
Methanotrophe Bakterien leben von Methan als einziger Kohlenstoffquelle. Im ersten Schritt der Methan-Verstoffwechselung wird dieses zu Methanol oxygeniert. Der diese Umsetzung ermöglichende Enzymkomplex, die membrangebundene Methanmonooxygenase (pMMO, „p“ für „particulate“), war hinsichtlich seiner Zusammensetzung lange unbekannt. Erst kürzlich bestätigte eine erste Strukturanalyse die Vermutung, dass im aktiven Zentrum Kupfer vorliegt, der Kenntnisstand ist jedoch sehr bruchstückhaft [pmmo_highlight_2010], [pmmo_2010]. Bei Kupfermangel bilden die Bakterien die erheblich besser untersuchte lösliche Methanmonooxygenase (sMMO), deren Hydroxylasekomponente zur Klasse der Oxidodieisen-Enzyme gehört. Die katalysierte Reaktion ist der formale Einschub eines O-Atoms eines O2-Moleküls in eine C–H-Bindung unter Reduktion des zweiten O-Atoms:
CH4 + NADH + H+ + O2 → CH3OH + NAD+ + H2O
Einen Überblick gibt [smmo_2011].
Gezeigt ist die Ruheform des Enzyms (die FeIIIFeIII-Form) sowie die reaktionsbereite reduzierte FeIIFeII-Form von Methylococcus-capsulatus-sMMOH in einer Auflösung von jeweils 2.1 Å (PDB-Code 1FZ1). MMOH ist mit ca. 360 Aminosäuren ein größeres Protein:
Das aktive Zentrum liegt ungewöhnlich tief im Inneren des durch zahlreiche α-Helix-Abschnitte verdichteten, für große und polare Substrate kaum zugänglichen Proteins. Die terminalen Liganden der FeIIIFeIII-Form sind beim linken Eisenatom 1 His, 1 Glu und ein Wassermolekül, beim rechten Fe-Zentrum 1 His und 2 Glu. Verbrückende Liganden sind 2 OH und 1 O,O'-Glu. Damit stehen der Ladung 6+ der beiden Eisen(III)-Atome die Ladung 6− auf der Seite der Liganden gegenüber – im hydrophoben aktiven Zentrum ist damit ein ionischer Zustand vermieden:
Das aktive Zentrum der reduzierten FeIIFeII-Form (PDB-Code 1FYZ) weist als terminale Liganden beim linken Eisenatom 1 His, 1 Glu und ein Wassermolekül, beim rechten Fe-Zentrum ebenfalls 1 His und 1 Glu auf. Verbrückende Liganden sind 1 H2O, 1 O,O'-Glu und ein weiteres Glu in einem ungewöhnlichen Bindungsmodus, nämlich als (μ-O),O'-Ligand; in diesem Bindungsmodus hat das verbrückende O-Atom die Rolle eines der beiden Hydroxido-Liganden der oxidierten Form übernommen. Auch das reduzierte Zentrum ist elektroneutral:
Die FeIIFeII-Form wird durch die Umsetzung mit einem Molekül O2 in die FeIV(μ-O)2FeIV-Form überführt. Der stabilste Spinzustand scheint antiferromagnetisch-high-spin zu sein. In diesem Zustand erfolgt die Methananlagerung an einen der μ-Oxido-Liganden. Ein möglicher Weg der Oxygenierung besteht dann in der Übertragung eines Methan-H-Atoms auf den μ-Oxido-Liganden unter Bildung eines FeIII(μ-OH)(μ-O)FeIV-Zentrums und anschließender Bindung des Methylradikals an den Oxido/Hydroxido-Liganden, wobei ein FeIII(μ-OH)(μ-OMe)FeIII-Fragment entsteht. Einen solchen „rebound“-Mechanismus werden wir beim Cytochrom P450 näher kennenlernen. Man beachte, dass für diesen Mechanismus mit einer hochreaktiven Oxidometall-Spezies keine freie Koordinationsstelle am Metallzentrum zu Beginn des Cyclus notwendig ist.
Bei der Klausur … : Bedeutung der Elektrostatik bei der Abschätzung des Redoxpotentials; Stabilisierung hoher Oxidationsstufen durch anionische Liganden.
[smmo_2011]
C. E. Tinberg, S. J. Lippard:
Dioxygen Activation in Soluble Methane Monooxygenase.
Acc. Chem. Res. 2011, 44, 280–288.
doi:
10.1021/ar1001473
[pmmo_highlight_2010]
R. A. Himes, K. Barnese, K. D. Karlin:
One is Lonely and Three is a Crowd: Two Coppers Are for Methane Oxidation.
Angew. Chem. Int. Ed. 2010, 49, 6714-6716.
doi:
10.1002/anie.201003403
[pmmo_2010]
R. Balasubramanian, S. M. Smith, S. Rawat, L. A. Yatsunyk, T. L. Stemmler, A. C. Rosenzweig:
Oxidation of methane by a biological dicopper centre.
Nature 2010, 465, 115–121.
doi:
10.1038/nature08992
[hr_2004]
M. Shoji, Y. Nishiyama, Y. Maruno, K. Koizumi, Y. Kitagawa, S. Yamanaka, T. Kawakami, M. Okumura, K. Yamaguchi:
Theory of chemical bonds in metalloenzymes I: Analytical and hybrid-DFT studies on oxo and hydroxo diiron cores.
Int. J. Quantum Chem 2004, 100, 887–906.
doi:
10.1002/qua.20286
Die bisher behandelten Enzymzentren haben eine Gemeinsamkeit: Als Liganden traten stets die Seitenketten von proteinogenen Aminosäuren auf. Lediglich bei einem Beispiel – der Urease – war eine solche Seitenkette geringfügig modifiziert. Da nur einige der 20 proteinogenen Aminosäuren geeignete funktionelle Gruppen tragen, ist in einem Protein die Zahl der metallbindenden Aminosäuren sehr beschränkt. Die wichtigsten Liganden sind:
Histidin ist unter den häufig vorkommenden Liganden (His, Asp, Glu, Cys, Tyr) der einzige Neutral-Ligand, alle übrigen sind Anionen. His stabilisiert daher als einziger Ligand nicht die höherwertige Stufe eines Redoxpaares. Das Potential eines Redox-Zentrums steigt daher mit der Zahl der His-Liganden. Der pKA-Wert des Histidins liegt bei 14.4, so dass gegenüber stark Lewis-sauren Metallzentren seitenketten-deprotoniertes His als zweizähniger, anionischer Ligand auftreten könnte. Dieser Bindungsmodus ist jedoch selten, im Rahmen dieser Vorlesung kommt er nur bei CuZnSOD vor.
Im einzähnigen Neutral-Ligand-Modus werden beide Tautomere gefunden (δ und ε beziehen sich auf den kürzesten Weg, auf dem man vom α-C-Atom aus das jeweilige N-Atom erreichen kann).
Neben der Funktion als Ligand findet sich nicht-metallgebundenes Histidin oft in der Rolle eines „proton shuttles“, der in einem Katalysecyclus Protonen leicht aufnehmen und abgeben kann. Der pKA-Wert des Histidinium-Kations von 6.0 zeigt, dass Histidin beim physiologischen pH weitgehend nicht-protoniert vorliegt, aber leicht Protonen von stärkeren Säuren als Wasser (pKA = 15.7) aufnehmen kann. Wäre Histidin nur geringfügig basischer, würde es im physiologischen Milieu als Kation vorliegen.
Die pKA-Werte von Asparaginsäure und Glutaminsäure betragen 3.9 und 4.1. Bei physiologischem pH liegen also auch ohne Metallunterstützung die Anionen vor.
Der pKA-Wert der Thiolfunktion des Cysteins ist mit 8.3 deutlich höher als bei den Carboxylfunktionen von Asp und Glu. Bei physiologischem pH liegt die Seitenkette daher in der neutralen, nicht-deprotonierten Form vor. Als Ligand tritt diese jedoch nicht auf. Durch die Bindung an die Lewis-sauren Zink-, Nickel(II), Eisen(II)- und Eisen(III)-Zentren – die wichtigsten Bindungspartner des Cysteins – wird das Cysteinat-Anion stabilisiert.
Tyrosin ist noch sauer genug, um nach Bindung an ein Lewis-acides Metallzentrum als deprotonierter Ligand aufzutreten (pKA(TyrH) = 10.1). Unter den hier zusammengestellten Seitenketten-Liganden erfüllt Tyrosinat am wenigsten die Forderung nach einem „unschuldigen“ Liganden (engl. innocent ligand oder spectator ligand), der das Zentralmetall bestenfalls durch seine Ladung beeinflusst. Tyrosin ist vielmehr leicht zum Tyrosyl-Radikal oxidierbar, das seinerseits als Ligand wirken kann. Tyrosin-Komplexe zählen daher potentiell zu einer Substanzklasse, die in der jüngsten Vergangenheit auch außerhalb der Bioanorganischen Chemie immer größeres Interesse auf sich zieht, den Komplexen mit radikalischen Liganden.
Methionin-Liganden kommen in den Cytochromen vor, wo sie die Koordinationssphäre von Häm-Eisen-Zentren vervollständigen sowie im Plastocyanin. Ansonsten ist dieser Thioether als Ligand bei weitem nicht so verbreitet wie die vorgenannten Aminosäure-Seitenketten.
Die Ligandeigenschaften dieser Seitenketten entsprechen denen geläufiger Liganden wie Pyridin oder Acetat. Die Chemie der von diesen Aminosäuren gebildeten aktiven Zentren erinnert daher stark an die Komplexchemie einfacher Werner-Komplexe. Die Modulation der Komplexeigenschaften erfolgt durch Variation unspektakulärer Parameter wie der Koordinationsgeometrie, der elektrostatischen Situation des Metalls oder der Nuclearität des Zentrums.
Eine neue Dimension wird bei den Häm-Zentren erschlossen. Porphyrine sind Liganden, die neben den bislang untersuchten high-spin-Systemen auch low-spin-Zustände stabilisieren können. In den zuerst behandelten Proteinen Myoglobin, Hämoglobin und Häm-Peroxidase ist der Porphyrin-Kern durch Methyl-, Vinyl- und Carboxyethyl-Reste substituiert. Die Formel zeigt dieses Protoporphyrin IX von der distalen Seite (in den gezeigten Proteinstrukturen von oben).
Im Hämoglobin des Blutes und im Myoglobin des Muskels nutzt die Natur einen high-spin-Eisen(II)–low-spin-Eisen(II)-Übergang, um das Metallzentrum zu einem Sauerstoff-Transporter zu machen. Bei einem high-spin–low-spin-Übergang geht es nicht um kleine Effekte, sondern es treten strukturell und energetisch drastische Veränderungen auf – es liegt ein molekularer Schalter vor.
In der Häm-Biochemie sind einige Abkürzungen üblich, die auch hier verwendet werden.
Ein geläufiges System bezeichnet mit Mb und Hb nur die Proteine im allgemeinen und definiert den jeweiligen Zustand durch vorangestellte Symbole. Die nicht-oxygenierte Eisen(II)-Form ist dann deoxyMb und deoxyHb. Durch die Anlagerung von Sauerstoff entsteht oxyMb und oxyHb. 1-e-Oxidation führt zu den nicht-O2-bindenden Eisen(III)-Formen metMb und metHb.
In diesem System lassen sich andere Addukte nicht gut darstellen. In Arbeiten, die sich mit der Anlagerung zum Beispiel von CO oder NO befassen, wird oft wie folgt abgekürzt: Aus deoxyMb und oxyMb wird Mb und MbO2, metMb bleibt erhalten. Die neuen Addukte heißen dann MbNO und MbCO, der Leser muss also wissen, dass Eisen(II) gemeint ist. Werden die Abkürzungsschemata vermischt, ist das Resultat meist selbsterklärend, lediglich bei NO-Komplexen ist Vorsicht geboten.
Der Weg des Sauerstoffs von der Lunge bis in die Mitochondrien beginnt mit der Bindung an Hämoglobin. In den Muskelzellen erfolgt die Übergabe an Myoglobin, das eine größere Bindungskonstante für O2 als Hb hat. Von diesem wird das Sauerstoffmolekül schließlich an Cytochrom-c-Oxidase (CcO) übertragen, bei dem die Sauerstoffaffinität den größten Wert erreicht. Im aktiven Zentrum von CcO, dem letzten Enzym der Atmungskette, wird Sauerstoff zu Wasser reduziert. Die Sauerstofftransportenzyme Hb und Mb kommen bei allen Wirbeltieren und vielen Wirbellosen vor. Das Mengenverhältnis von Mb zu Hb ist zwischen den Organismen sehr verschieden. Besonders große Mb-Mengen werden von im Wasser lebenden lungenatmenden Tieren genutzt, um viel Sauerstoff für lange Tauchgänge speichern zu können. So können Wale und Robben aufgrund ihres hohen Mb-Spiegels ca. ½ h unter Wasser bleiben. Das in der Biochemie allgegenwärtige Pottwal-Mb (engl. sperm whale myoglobin) erklärt sich aus der hohen Verfügbarkeit dieses Proteins.
Beide Transportenzyme sind nur dann in der Lage Sauerstoff zu binden, wenn das Eisenzentrum in der Oxidationsstufe +II vorliegt (deoxyMb und deoxyHb). Oxidation zur dreiwertigen Stufe unter Bildung von metMb oder metHb führt zur Desaktivierung.
Eine neu entdeckte Funktion von Enzymen der Mb- und Hb-Familie scheint die Beseitigung von NO zu sein. Mbs and Hbs können NO zu Nitrat oxidieren. Ausgehend von oxyMb oder oxyHb, deren FeII-O2-Fragment sich hierbei wie eine FeIII-O2•−-Funktion verhält (siehe auch weiter unten), lässt sich die Umsetzung gemäß
NO + O2•− = NO3−
formulieren, wobei metMb oder metHb zurückbleibt und anschließend durch eine 1-e-Reduktion wieder in den zweiwertigen Ausgangszustand überführt werden muss.
Häm-Proteine traten in der Evolution weit vor dem Aufkommen von Sauerstoff in der Atmosphäre auf. Die NO-bindenden Eigenschaften haben zu der Hypothese geführt, dass das „Ur-Hämoglobin“, dessen Entstehung vor ca. 3.5 Milliarden Jahren vermutet wird, dem NO-Stoffwechsel diente und dass der O2-Transport eine erst später evolvierte Eigenschaft ist.
Mb ist ein 153 Aminosäuren langes Protein, das durch acht α-Helices dominiert ist. Eine hydrophobe Tasche des Apoproteins enthält einen Eisen(II)-Protoporphyrin-IX-Komplex (Häm b) ohne kovalente Anbindung des Porphyrins nur über einen His-Fe-Kontakt (siehe Abbildung bei oxyMb). Das unmittelbar an Eisen gebundene His ist das „proximale“ His (His93, oft als F8 adressiert [8. Aminosäure auf α-Helix F]). Auf der anderen Häm-Seite liegt die O2-Bindungsstelle. Hier wird in der Regel ein weiteres His gefunden, das „distale“ His (His64 oder E7), das in passender Entfernung zum Häm lokalisiert ist, um als N-H-Donor eine Wasserstoffbrückenbindung zu H-Brücken-Akzeptoren aufbauen zu können.
Das Bild zeigt deoxy-Mb bei hoher Auflösung (1 Å), PDB-Code 1A6N. Das distale His wird fehlgeordnet gefunden, das auf der distalen Seite eingezeichnete Wassermolekül ist in unterbesetzter Lage. Aufgrund der für Wasserstoffbrückenbindungen typischen N···O-Abstände zwischen der Wasserlage und dem distalen His (2.76 Å) sowie der Wasserlage und einem der vier Porphyrin-Stickstoffatome (2.74 Å) lässt sich die Fehlordnung des distalen His verstehen: in einem Teil der den Kristall bildenden Mb-Moleküle fehlt das einzelne Wassermolekül auf der distalen Seite. Dies ist verständlich, da es außer den beiden N···O-Kontakten keine weiteren Bindungsmöglichkeiten für den Wasser-Dipol gibt – der distale Hohlraum ist außer von His nur von hydrophoben Seitenketten ausgekleidet (siehe unten). Das distale His nimmt die linke Position aber nur bei Anwesenheit von Wasser ein. Fehlt das Wassermolekül, bewegt sich das distale His nach rechts in Richtung auf den Proteinrand, wo es einen nicht gezeigten Kontakt zu einem Wassermolekül außerhalb der O2-Bindungstasche aufbauen kann.
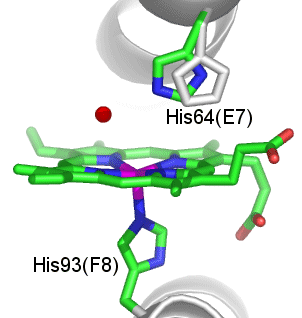
Das Eisen(II)-Zentrum in deoxyMb liegt im S=2-Grundzustand vor, es ist ein high-spin-Zentrum.
Auch die Struktur von oxyMb liegt in hoher Auflösung vor (PDB-Code 1A6M). Der Blick auf das Holoenzym zeigt die Lage des Häms in einer von α-Helices gebildeten Tasche. Die α-Helices sind in der üblichen Weise bezeichnet:
Im aktiven Zentrum ist ein Sauerstoffmolekül gebunden, dessen weitere Umgebung mit Ausnahme des distalen His nur aus hydrophoben Seitenketten besteht:
Die Wechselwirkung zwischen dem distalen His und O2 wird deutlicher, wenn der Betrachter von der linken Seite in das aktive Zentrum hineinschaut:
Das distale His ist Donor einer Wasserstoffbrückenbindung zum terminalen O-Atom des O2-Liganden. Die N–H···O-Bindung ist nicht die Ursache der gewinkelten Fe-O-O-Anordnung, sie nutzt diese jedoch zum Aufbau der zusätzlichen Wechselwirkung und erhöht so die Bindungskonstante für O2.
oxyMb liegt im Singulett-Grundzustand vor, was Pauling zu der Schlussfolgerung veranlasste, oxyMn sei ein low-spin-Eisen(II)-Komplex.
Die Stabilisierung des O2-Komplexes durch die H-Brückenbindung zum distalen His scheint der wesentliche Grund für die relativ geringe CO-Affinität von Mb und Hb zu sein. Während isoliertes Häm CO ca. 105 mal wirksamer bindet als O2, so ist dieses Verhältnis bei Mb und Hb um den Faktor 1000 geringer. Eine dann 100-fache Affinität zu CO weist Kohlenmonoxid immer noch als giftiges Gas aus, wenn es in unphysiologisch hoher Konzentration eingeatmet wird. Die zum Beispiel beim Häm-Abbau entstehende geringe physiologisch gebildete CO-Menge jedoch verliert bei diesem Verhältnis der Bindungskonstanten die Fähigkeit, Mb oder Hb zu blockieren.
Es liegen zahlreiche Strukturanalysen zur Bindung von CO an Mb vor. Die Leu29Trp-Mutante von Pottwal-Mb, bei welcher der hydrophobe Raum auf der distalen Seite durch die Mutation verkleinert ist, wurde detailliert untersucht. Das CO-Addukt (PDB-Code 1DO1) zeigt ein nur geringfügig abgewinkeltes Fe-C-O-Fragment:
Im Vergleich mit dem oxyMb-Zentrum ist das distale His nun nach rechts gedrängt, die Ausrichtung einer N-H-Funktion auf CO ist nicht gegeben. Lange Zeit wurden die beiden letzten Bilder so interpretiert, dass CO das distale His unter nennenswertem Energieaufwand zur Seite schiebt, woraus eine verminderte Bindungskonstante resultiert hätte. Aktuelle Untersuchungen haben diese Vorstellung nicht bestätigt. Das terminale O-Atom von O2 scheint im Vergleich mit dem O-Atom des Kohlenmonoxids vielmehr der deutlich bessere H-Brückenbindungs-Akzeptor zu sein. Die Bindung zu CO ist also nicht durch sterische Belastung besonders destabilisiert, die Bindung zu O2 ist vielmehr besonders stabil. Dieses Ergebnis legt nahe, dass die Natur eine höhere negative Beladung des terminalen Atoms des O2-Liganden gegenüber dem CO-Sauerstoffatom für eine starke H-Brückenbindung nutzt. Diese Schlussfolgerung leitet zu einem Vergleich der Bindungsverhältnisse in oxyMb und MbCO über, die eine Gemeinsamkeit verbindet: beide Komplexe liegen im Singulett-Grundzustand vor.
Die O2-Addukte oxyMb und oxyHb sind diamagnetisch. Die Formulierung eines Häm-O2-Komplexes mit einem S=0-Grundzustand bereitet Probleme. Der auf Pauling zurückgehende älteste Versuch einer Deutung sieht oxyMb und oxyHb als low-spin-Eisen(II)-Zentren, an die ein Singulett-O2-Ligand koordiniert. Die Kontroverse um diese Formulierung der Häm-O2-Bindung ist alt. Weiss schlug als Alternative zur Annahme von Singulett-Sauerstoff einen Hyperoxido-low-spin-Eisen(III)-Komplex vor. Die Annahme eines anionischen Liganden würde dabei das Verhalten als H-Brückenbindungs-Akzeptor erklären. Zur Deutung des Singulett-Grundzustandes muss eine antiferromagnetische Kopplung des Superoxid-Spins und des low-spin-Eisen(III)-Spins gefordert werden.
DFT-Rechnungen favorisieren ein FeIII–2O2•−. Weiss' Vorstellung hat danach den größten Anteil an der realen Situation, nicht zutreffend ist jedoch eine zu ionische Formulierung. In Populationsanalysen ergeben sich als Ladung des O2-Liganden Werte um −0.2 e− [heme_diatomicsbinding_2005].
Multikonfigurationsmethoden geben einem FeII–1O2ein höheres Gewicht. Rechnungen mit ab-initio-Methoden außerhalb der Dichtefunktionaltheorie tendieren zu einem höheren Gewicht von Paulings Formulierung. So ergibt die Anwendung von Multikonfigurationsmethoden ungefähr gleiche Anteile von Weiss' und Paulings Formel [heme_o2binding_2005]. Im Orbitalbild lassen sich die Unterschiede zwischen den Betrachtungsweisen aufzeigen. Hierbei wird das folgende Koordinatensystem benutzt, das zur üblichen Benennung der Metall-d-Orbitale in oktaedrischem Feld führt (man beachte, dass in der Literatur bisweilen die x- und y-Achse gegenüber der Abbildung um 45° um z gedreht sind, so dass die Orbitalbezeichnungen andere sind; vor allem sind xy und x2−y2 vertauscht). Die Fe-O-O-Ebene enthält in guter Näherung die z-Achse und die Winkelhalbierende zwischen x und y:
Die O2-Koordination lässt sich in Einzelschritten betrachten:
1. Ausgangspunkt ist ein high-spin-Eisen(II)-Ion, für welches das Kristallfeldmodell die d-Orbital-Reihenfolge (xz,yz), xy, z2, x2−y2 ergibt. Der Porphyrin-Ligand wird hier als stärker feldaufspaltend eingestuft als der Mittelwert aus einem His-Liganden und einer unbesetzten Koordinationsstelle. Durch Hinzufügen von π-Wechselwirkungen im Häm ergibt sich xy, (xz,yz), z2, x2−y2.
2. Ein Triplett-Sauerstoff-Diradikal, das sich in der experimentell bestimmten Schrägstellung entlang der z-Achse nähert, bindet mit lokaler σ-Symmetrie in das z2-Orbital des Metalls. O2 benutzt hierzu eines seiner halbbesetzten antibindenden Grenzorbitale.
3. Durch den zusätzlichen Liganden kommt es am Eisen(II)-Zentrum zur Schaltung von high-spin zu low-spin. Zugleich verringert sich die tetragonale Verzerrung des Ligandenfelds. Die Orbitalreihenfolge wird dabei erhalten bleiben, da ein Porphyrin-Ligand zu einer höheren Feldaufspaltung führt als das His/O2-Paar.
4. Es gibt noch zwei ungepaarte Spins. Der eine in (xz,yz)3, der andere im senkrecht zur Fe-O-O-Ebene stehenden O2-π*-Orbital. Durch Linearkombination lässt sich ein spintragendes Orbital der (xz,yz)3-Gruppe so zum antibindenden Sauerstofforbital anordnen, das eine Fe-O-Bindung mit lokaler π-Symmetrie entsteht.
5. Für die Orbitalanordnung mit lokaler π-Symmetrie ist antiferromagnetische Kopplung zu erwarten. Wird an diesem Punkt das Elektronenpaar der Eisen-Sauerstoff-σ-Bindung den Konventionen entsprechend dem elektronegativeren Sauerstoff zugerechnet, so stellt die jetzt erhaltene Konfiguration den Weisschen FeIII–2O2•−-Zustand dar.
6. Wird stattdessen das spingekoppelte Elektronenpaar ganz dem Eisen zugeordnet, ergibt sich Paulings Konfiguration. Aktuelle DFT-Rechnungen stützen Weiss' Formel, indem sie überschüssige α-Spindichte entsprechend 1 Elektron in der (xz,yz)-Linearkombination sehen. Zusätzlich liegt im passenden Sauerstoff-π*-Orbital β-Spin vor. Andere quantenchemische Methoden finden die Spintrennung in der Eisen-Sauerstoff-π-Bindung als weniger ausgeprägt.
Der Vorteil der FeIII–2O2•−-Formulierung wird aus dieser Betrachtung deutlich. Es ist kein spinverbotener 3O2→1O2-Übergang enthalten. Spinpaarung beschränkt sich auf den high-spin-low-spin-Schaltvorgang am Zentralmetall.
Die Formulierung von oxyMb als Komplex mit einem anionischen Liganden erleichert es, die Stabilisierung der O2-Anbindung durch die H-Brücke vom distalen His einzuordnen. Der H-Brücke werden zwei wesentliche Funktionen zugeschrieben: (1) sie behindert die Autoxidation von Häm durch Sauerstoff, (2) sie ist hauptverantwortlich für die Diskriminierung zwischen O2 und CO. Letzteres liegt als Neutralligand vor, dessen Donor-Akzeptor-Verhalten zu einer geringeren Beladung und damit H-Brücken-Akzeptor-Fähigkeit des Sauerstoffatoms führt.
Reversible O2-Bindung benötigt mehr als ein isoliertes Häm, dessen Umsetzung mit O2 zu oxidierten Formen führt. Mechanistische Voraussetzung für eine schnelle und quantitative Oxidation ist die Möglichkeit der Dimerenbildung. In „picket-fence-Porphyrinen“ und deren Analogen (Figure 3 in [heme_models_2004]) ist die Dimerisierung verhindert, so dass hier die stereoelektronischen Voraussetzung für reversible O2-Bindung vorliegt.
Mb ist nur in der Eisen(II)-Form zur O2-Bindung (und CO-Bindung) in der Lage. Die Eisen(III)-Form, met-Myoglobin, bindet nur an polare Liganden. Kommt es ohne die Anwesenheit solcher Liganden wie Cyanid oder Azid zur Oxidation, so wird ein distaler Aqua-Ligand gefunden (PDB-Code 1BZ6):
Die Ausrichtung des distalen Histidins und ein kurzer N···O-Abstand zeigen eine Wasserstoffbrückenbindung an, wahrscheinschlich vom Typ HO–H···N.
Für den Zutritt von O2, CO oder NO zum Häm-Zentrum von Mb oder Hb ist kein Diffusionskanal zu erkennen. Das aktive Zentrum ist vielmehr nach außen abgeschirmt. Um die zweiatomigen Substrate in das Enzyminnere zu transportieren, sind Bewegungen des Enzyms notwendig. Diese werden durch Hohlräume ermöglicht, die durch Strukturanalysen nach Xenonbeladung sichtbar gemacht werden können. Die Wanderung vor allem von CO und NO unter gleichzeitiger Bewegung des Proteins kann durch zeitaufgelöste Laue-Kristallographie verfolgt werden. Hierzu wird die Struktur durch kurze Röntgenpulse abgefragt, nachdem die Eisen-Ligand-Bindung photolysiert wurde [heme_dynamics_2004].
| Mb | X | Fe-Npor | Fe-Hisprox | Fe-X | X-Hisdist | PDB |
|---|---|---|---|---|---|---|
| deoxy | – | 2.07 | 2.14 | 2.76 | 1A6N | |
| oxy | O2 | 2.00 | 2.06 | 1.81 | 2.67 (zu Oterm) | 1A6M |
| met | H2O | 2.04 | 2.14 | 2.13 | 2.67 | 1A6K |
| met | H2O | 2.03 | 2.16 | 2.17 | 2.69 | 1BZ6 |
| nitrosyl | NO | 1.97 | 2.31 | 1.86 | 2.98 (O) 3.23 (N) |
1JDO |
| carbonyl | CO | 2.00 | 2.11 | 1.73 | 3.21 | 1BZR |
Bei der Klausur … : Voraussetzungen für den high-spin–low-spin-Übergang; Auswirkung des Übergangs auf die Metall-Ligand-Bindungsordnung und die Kovalenz der Bindung; Aufspaltung der d-Niveaus in Porphyrin-Komplexen, räumliche Lage der Grenzorbitale in geläufigen zweiatomigen Liganden.
[heme_o2binding_2005]
K. P. Jensen, B. O. Roos, U. Ryde:
O2-binding to heme: electronic structure and spectrum of oxyheme, studied by multiconfigurational methods.
J. Inorg. Biochem. 2005, 99, 45–54.
doi:
10.1016/j.jinorgbio.2004.11.008
[heme_diatomicsbinding_2005]
L. M. Blomberg, M. R. A. Blomberg, P. E. M. Siegbahn:
A theoretical study on the binding of O2, NO and CO to heme proteins.
J. Inorg. Biochem. 2005, 99, 949–958.
doi:
10.1016/j.jinorgbio.2005.02.014
[heme_models_2004]
J. P. Collman, R. Boulatov, C. J. Sunderland, L. Fu:
Functional Analogues of Cytochrome c Oxidase, Myoglobin, and Hemoglobin.
Chem. Rev. 2004, 104, 561–588.
doi:
10.1021/cr0206059
[heme_dynamics_2004]
M. Brunori, D. Bourgeois, B. Vallone:
The structural dynamics of myoglobin.
J. Struct. Biol. 2004, 147, 223–234.
doi:
10.1016/j.jsb.2004.04.008
Wird Sauerstoff als Hyperoxido-Ligand gebunden, fungiert O2 also nicht nur als Lewis-Base, sondern bringt Redox-Aktivität ins Spiel, wird von einem „nicht-unschuldigen Ligand“ gesprochen (engl. non-innocent ligand). Der wohl typischste nicht-unschuldige Ligand ist Stickstoffmonoxid, NO. Dessen IUPAC-Formulierung als Dreielektronen-Donor trifft den Bindungsmodus in Enzymzentren wohl am schlechtesten.
Unter den 2-atomigen Liganden mit physiologischer Funktion, O2, CO und NO, nimmt Stickstoffmonoxid eine Sonderstellung ein. NO ist der einzige Ligand, der sowohl an deoxyMb/Hb als auch an metMb/Hb binden kann. Die Bindung zum Eisen(II)-Zentrum ist stabiler, zugleich bindet NO deutlich stärker an dieses Zentrum als Sauerstoff und etwa gleich stark wie CO (Bindungsdissoziationsenergien an Imidazol/Häm: O2 63, CO 146, NO 151 kJ mol−1). Die Präferenz für Eisen(II) wird beim Vergleich der freien Bindungsenergien für NO deutlich: deoxyMb −64, metMb ca. −20 kJ mol−1).
Die Strukturanalyse am stabileren Eisen(II)-Komplex zeigt gewinkelten Aufbau (PDB-Code 1JDO). Das distale His64 bildet wie im O2-Addukt eine Wasserstoffbrückenbindung zum terminalen Atom des zweiatomigen Liganden:
Die Bindungsmöglichkeiten von NO-Liganden sind ungewöhnlich vielfältig. Formale NO+-Liganden sind isoster zu CO und CN−, der neutrale NO-Ligand ist ein Radikal und ein formaler NO−-Ligand ist isoster mit O2 und kann wie dieses als Singulett- oder als Triplett-Spezies an ein Metallzentrum binden.
Zur Systematisierung der jeweiligen elektronischen Situation wird die nach Enemark und Feltham benannte Nomenklatur verwendet. Hierzu wird über die Metall-Valenzelektronen und die antibindenden NO-Elektronen summiert. Reagiert zum Beispiel wie in MbNO Stickstoffmonoxid (1 Elektron in π*) mit deoxyMb (FeII = d6), so ergibt sich der {FeNO}7-Fall. Typisch für diese Elektronenzahl ist eine gewinkelte Fe-N-O-Gruppe, wobei der Bindungswinkel am Stickstoffatom typischerweise ca. 140° beträgt. Zur Fe-NO-Bindung in MbNO liegt eine Arbeit vor [heme_no_2004], welche die ungepaarte Spindichte des {FeNO}7 dem Eisenatom zuordnet. Von den Möglichkeiten, die zu einem S=½-Grundzustand führen (low-spin-FeII + NO-Radikal oder low-spin-FeIII + Singulett-NO− oder low-spin-FeIII + Triplett-NO− mit antiferromagnetischer Kopplung) wird die zweite Möglichkeit gefunden, bei der ein low-spin-FeIII-Häm-Fragment einen Singulett-NO−-Liganden bindet (rechte Formel):
Nitrophorine sind NO-bindende Eisen(III)-Häm-Enzyme. Die im Vergleich mit Häm-Eisen(II)-Zentren nicht so starke NO-Anbindung dient dem Zweck der Nitrophorine: blutsaugende Insekten injizieren diese Verbindungen mit ihrem Biss in die Blutbahn des Opfers. Die Nitrophorine setzen NO frei; dieses dient als gefäßerweiterndes Hormon und den Insekten steht mehr Blut zur Verfügung.
Ein ungewöhnlicher Bindungsmechanismus für zwei NO-Moleküle zeigt ein Nitrophorin aus der Gattung Cimex (PDB-Code 1Y21):
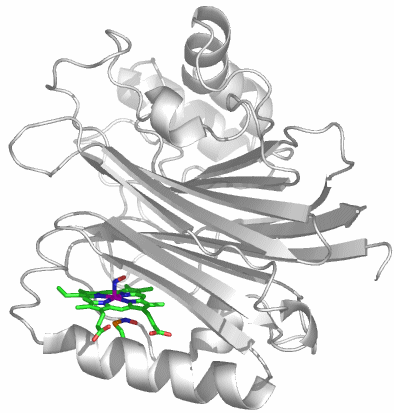
Das aktive Zentrum zeigt einen nitrosylierten Cys-Ligand, der nach der Reaktion des ersten NO-Moleküls mit dem Eisen(III)-Zentrum gebildet wird:
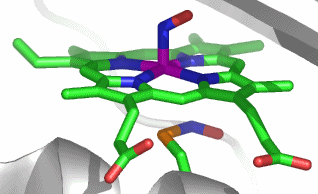
Man beachte die Stabilisierung der dreiwertigen Stufe durch einen Cysteinato-Ligand. Ein Vorschlag für den Mechanismus der reversiblen NO-Bindung ist in [np_2005] gegeben.
Die bindungstheoretische Beschreibung von Häm-{FeNO}6-Zentren ist Gegenstand der Diskussion. Die naheliegende Formulierung als low-spin-Eisen(II)-NO+-Fragment deutet auch hier die vorliegenden spektroskopischen Daten nicht befriedigend (in der Abbildung eingeklammert), so dass ein low-spin-Eisen(III)-NO-Zustand diskutiert wird (im Bild rechts) [np_bonding_2005]:
Das endogen als Hormon gebildete oder durch Insekten eingebrachte NO führt zur Gefäßerweiterung, indem es an ein Rezeptorenzym anbindet, der löslichen Guanylat-Cyclase, die Guanosintriphosphat (GTP) zu cyclischem Guanosinmonophosphat (cGMP) umwandelt; cGMP trägt dann den Reiz weiter. Lösliche GC gehört zu den Schlüsselenzymen des Herz-Kreislauf-Systems. Trotzdem liegt Strukturinformation nur für verwandte Proteine vor, die darüberhinaus einen besonders originellen Bindungszustand nicht zeigen: Es wird angenommen, dass in sGC ein low-spin-Eisen(II)-Häm-Zentrum mit einem proximalen His durch die Anlagerung von NO auf der distalen Seite die Bindung zum proximalen His aufgibt und so den Schaltreiz auslöst.
Bei der Klausur … : noch einmal: Unterschiede zwischen wichtigen zweiatomigen Liganden mit deren Grenzorbitalen verknüpfen: CO, CN−, NO+, NO, NO−, O2, O2•−, O22−.
[np_bonding_2005]
F. A. Walker:
Nitric oxide interaction with insect nitrophorins and thoughts on the electron configuration of the FeNO 6 complex.
J. Inorg. Biochem. 2005, 99, 216–236.
doi:
10.1016/j.jinorgbio.2004.10.009
[np_2005]
A. Weichsel, E. M. Maes, J. F. Andersen, J. G. Valenzuela, T. K. Shokhireva, F. A. Walker, W. R. Montfort:
Heme-assisted S-nitrosation of a proximal thiolate in a nitric oxide transport protein.
PNAS 2005, 102, 594–599.
doi:
10.1073/pnas.0406549102
[heme_no_2003]
Y. Zhang, W. Gossman, E. Oldfield:
A Density Functional Theory Investigation of Fe-N-O Bonding in Heme Proteins and Model Systems.
J. Am. Chem. Soc. 2003, 125, 16387–16396.
doi:
10.1021/ja030340v
Die Reaktivität eines Häm-Zentrums lässt sich durch die Wahl der zusätzlichen Liganden steuern. In Cytochrom c blockieren zwei weitere Liganden das Eisenatom für eine Substratbindung, so dass nur der Elektronenausstausch möglich wird. Bei der Häm-Katalase und beim Cytochrom P450 führt ein anionischer Ligand zur Stabilisierung höherer Oxidationsstufen als bei Mb und Hb möglich. Besonders lehrreich ist die Reaktionssteuerung bei der C-H-Aktivierung durch Cytochrom P450. Die Annäherung eines hydrophoben Substrats, das selbst nicht zum Ligand wird, lässt eine Reaktionskaskade ablaufen, an deren Ende wie bei MMO eine C–H-Bindung oxygeniert worden ist. Die Natur zeigt hier beim Start der Kaskade, wie raffiniert ein molekularer Schalter konstruiert sein kann.
Cytochrome, denen aufgrund der Sechsfachkoordination ihrer Häm-Eisen-Zentren durch zwei His- und/oder Met-Liganden jede Gelegenheit genommen ist, einen Prozess zu katalysieren, bei dem sich zuerst ein Substrat anlagern muss, dienen wie die Ferredoxine dem 1-e-Transfer, jedoch bei mittleren Potentialwerten. Cytochrom c ist ein ubiquitäter 1-Elektron-Überträger, dessen elektrochemisches Potential ca. ½ V höher ist als das der Ferredoxine.
Es liegen Kristallstrukturanalysen an reduziertem (PDB-Code 1YCC, Auflösung 1.2 Å) und oxidiertem (PDB-Code 2YCC, Auflösung 1.9 Å) Hefe-Cytochrom c vor.
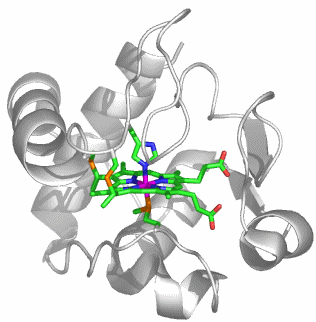
Das Häm-Molekül ist nicht wie bei den bisher behandelten Enzymen nicht-kovalent mit dem Apoprotein verbunden, vielmehr sind zwei Cys-Reste an die beiden Vinylsubstituenten des Protopophyrin IX addiert. Im Komplex mit Eisen heißt die prosthetische Gruppe nun „Häm c“:

Cytochrome teilen mit Ferredoxinen die Eigenschaft, in ihrem charakteristischen Potentialbereich die Weitergabe eines Elektrons mit niedriger Reorganisationsenergie zu leisten. Die folgende Übersicht über die Abstände in den beiden Oxidationszuständen zeigt eine mittlere Änderung von nur ca. 0.03 Å:
Übersicht über Fe-Ligand-Abstände in Å:
| ls-FeII | ls-FeIII | |
|---|---|---|
| Fe-Por | 2.01 | 1.99 |
| Fe-NHis | 2.01 | 1.99 |
| Fe-SMet | 2.43 | 2.35 |
Man vergleiche hiermit die Radiendifferenz für oktaedrisch koordiniertes Eisen(II) und Eisen(III): high-spin 0.14, low-spin 0.06 Å. Es wird deutlich, dass ein elektronenleitendes Hämenzym nur in den Kombinationen ls-FeII/ls-FeIII oder ls-FeII/hs-FeIII die notwendige kleine Reorganisationsenergie möglich macht.
Häm-Katalase dient der Disproportionierung von H2O2, das zum Beispiel von SODs produziert wird:
H2O2 = H2O + ½ O2
Bei der Häm-Katalase ist die biologische Funktionseinheit wahrscheinlich das Tetramer der abgebildeten asymmetrischen Einheit (PDB-Code 1QWL, 1.6 Å Auflösung):
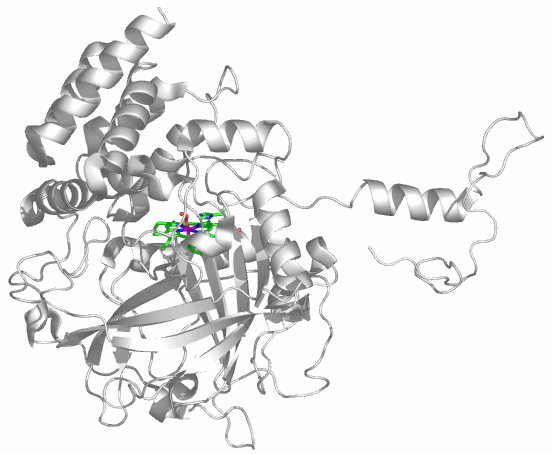
Die Proteinstruktur hat wenig Ähnlichkeit mit Myoglobin und doch ist das aktive Zentrum eine verblüffend konservative Variation des Mb-Zentrums. Der Cofaktor des Apoproteins ist ein Häm b. Oberhalb des Häms befindet sich wie in Mb und Hb ein vor allem durch Phenylalanin-Seitenketten hydrophob ausgekleideter Raum, der auch ein „distales“ His enthält. In dessen Umgebung findet sich ein Wassermolekül in einem N···O-Abstand von 2.65 Å. 2.42 Å vom Wassermolekül entfernt ist ein O-Atom eines an Eisen(III)(?) gebundenen HO2−-Liganden. Der auffälligste Unterschied zu metMb ist der Ersatz des proximalen His durch einen Tyrosinato-Ligand, der die dreiwertige Stufe des Eisens als reduzierte Form des Redoxpaares FeIII/FeIVPor•+ stabilisiert.
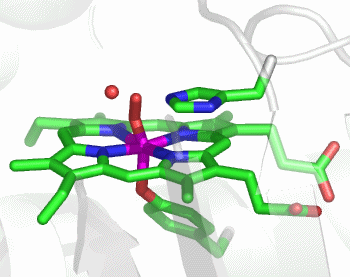
Die H2O2-Disproportionierung erfordert einen 2-Elektronen-Redoxprozess. Im ersten Schritt wird die FeIII-Ruheform des Enzyms um zwei Elektronen oxidert, es entsteht eine Oxidoeisen(IV)-Porphyrinradikalkation-Spezies, die anschließend wieder in die Ruheform zurückkehrt:
Por-FeIII + H2O2 → Por•+-FeIV=O + H2O
Por•+-FeIV=O + H2O2 → Por-FeIII + H2O + O2
Die Monooxygenasen der Cytochrom-P450-Familie (benannt nach λmax des CO-Addukts) sind ubiquitär und gehören zu den wichtigsten Enzymen des Metabolismus hydrophober körperfremder Stoffe und der Modifikation hydrophober Hormone, der Steroide. In [p450_2005] wird geschätzt, dass ca. die Hälfte aller derzeitiger Arzneistoffe durch ein einziges Cytochrom-P450-Enzym der Leber hydroxyliert wird. Cytochrom-P450-Enzyme vermitteln Insekten Resistenz gegen Insektizide und Pflanzen Resistenz gegen Herbizide.
Die katalysierte Reaktion ist
RH + O2 + 2 H+ + 2 e− = ROH + H2O
Die beiden Elektronen werden von Ferredoxinen bereitgestellt; im unten gezeigten Fall von P450cam aus Pseudomonas putida von Putidaredoxin.
Die Abbildung zeigt die Campher-5-monooxygenase P450cam aus Pseudomonas putida bei 1.9 Å Auflösung in der Campher-beladenen Form (PDB-Code 1T86, im Bild des Holoenzyms ist Campher rot dargestellt):
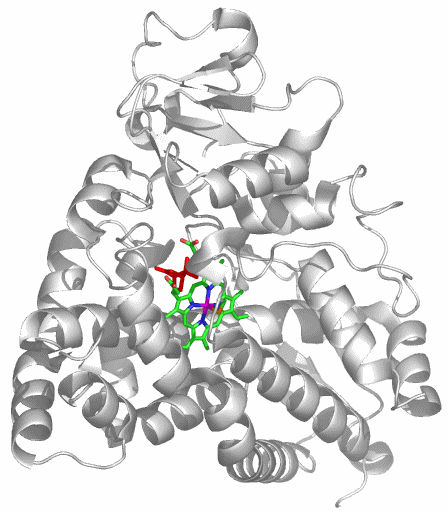
Im aktiven Zentrum ist auf der proximalen Seite ein Cysteinato-Ligand zu erkennen. Wie schon bei der Häm-Katalase liegt also ein anionischer Ligand vor, der höhere Oxidationsstufen des Eisenzentrums stabilisiert. Der Substrat-Bindungsbereich auf der distalen Seite ist hydrophob ausgekleidet und entsprechend der voluminösen Substrate recht groß.
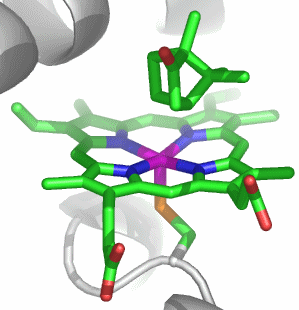
Die leichte Zugänglichkeit des ausgedehnten Substratbindungsbereichs und die hohe Reaktivität des zur C-H-Aktivierung fähigen aktiven Zentrums verlangen nach einem Schaltmechanismus, der die Kontrolle der Reaktivität sicherstellt. Der aus [p450_2005] entnommenene Katalysecyclus zeigt die wesentlichen Elemente dieses kontrollierten Umgangs mit einem aggressiv reaktiven Zentrum. Zugleich zeigt er beispielhaft, wie die Natur eine typisch „technische“ Aufgabe löst. Es geht um die grundlegende Frage des Sensorbaus, wie die An- oder Abwesenheit eines unreaktiven Moleküls in einen elektrischen Impuls umgesetzt werden kann:
Der Katalysecyclus startet bei der Ruheform des Enzyms, in der die Koordinationszahl eines Eisen(III)-Zentrums durch einen Aqua-Liganden zu sechs ergänzt ist. Im Einklang mit dieser Ligandausstattung ist der Spinzustand des Zentrums low-spin. Das elektrochemische Potential liegt typischerweise unter −0.3 V und damit unter dem Potential des als Reduktionsmittels bereitstehenden Putidaredoxins (E0' ca. −0.2 V), so dass keine Reaktion eintritt, die Eisen(II) erzeugen würde und damit den Angriff von O2 einleiten würde.
Schritt a: ein hydrophobes Substrat, das aufgrund des hydrophoben Effekts in der Enzymtasche bindet, verdrängt den Aqua-Ligand vom Eisenzentrum. Als Folge der Erniedrigung der Koordinationszahl kommt es zum low-spin→high-spin-Übergang – dem erwarteten Schaltprozess. Als Folge der Änderung von Spinzustand und Koordinationszahl steigt das elektrochemische Potential auf ca. −0.17 V und damit über den Wert des Ferredoxins.
Schritt b: Aufgrund der Umkehr der Potentialabfolge kommt es nun zur Reduktion des Hämzentrums durch das Ferredoxin. Wegen der Koordinationszahl 5 entsteht high-spin-Eisen(II).
Schritt c: high-spin-Eisen(II) ist in der Lage, O2 anzulagern. In Analogie zu Mb und Hb wird das Addukt als Eisen(III)-Hyperoxido-Fragment formuliert – eine Formulierung, die wegen des Eisen(III)-stabilisierenden Cysteinato-Liganden als weniger problematisch erscheint als bei den O2-Transportenzymen.
Schritt d: Es kommt erneut zur Reduktion. Das zweite Elektron der Bruttogleichung ist damit in den Prozess eingetreten. Das aktive Zentrum liegt nun als low-spin-Peroxido-eisen(III)-Fragment vor.
Schritt e: Es fehlen noch die beiden Protonen. Das erste bindet an die basischste Position, das terminale O-Atom des Peroxido-Liganden, der dadurch zum Hydroperoxido-Anion wird.
Schritt f: Durch das zweite Proton wird die gute Abgangsgruppe Wasser gebildet. Nach dessen Abspaltung reduziert das FeIIIPor-Zentrum das 6-Elektronen-Sauerstoffatom zu einem terminalen Oxido-Ligand. Bei diesem 2-Elektronenschritt wird es selbst zu einem FeIVPor•+ oxidiert. (Man vergleiche mit dem analogen Vorgang bei der Häm-Katalase.)
Schritt g: Es folgt der komplexeste Einzelschritt, für den Analogie zur C-H-Aktivierung bei der löslichen Methan-Monooxygenase angenommen werden kann: Übertragung des CH-Wasserstoffatoms unter Protonierung des terminalen Oxido-Liganden und 1-Elektronen-Reduktion des Häms. Anschließend Hydroxylierung des Alkyl-Radikals nach dem Rebound-Mechanismus unter Übertragung eines weiteren Elektrons auf das Häm.
Schritt h: Nach der Abspaltung des monooxygenierten Substrats stellt die Anlagerung eines Aqua-Liganden wieder die Ruheform her.
[p450_2011]
C. Jung:
The mystery of cytochrome P450 Compound I: A mini-review dedicated to Klaus Ruckpaul.
Biochimica et Biophysica Acta (BBA) - Proteins & Proteomics 2011, 1814, 46–57.
doi:
10.1016/j.bbapap.2010.06.007
[p450_2010]
S. Shaik, S. Cohen, Y. Wang, H. Chen, D. Kumar, W. Thiel:
P450 Enzymes: Their Structure, Reactivity, and Selectivity—Modeled by QM/MM Calculations.
Chem. Rev. 2010, 110, 949–1017.
doi:
10.1021/cr900121s
[p450_2005]
I. G. Denisov, T. M. Makris, S. G. Sligar, I. Schlichting:
Structure and Chemistry of Cytochrome P450.
Chem. Rev. 2005, 105, 2253–2278.
doi:
10.1021/cr0307143
Bei Plastocyanin und den CuA-Zentren geht es erneut um die 1-e-Übertragung, nun aber bei hohem Potential – bei viel höherem Potential, als es das Standardpotential des Kupfer(I,II)-Redoxpaares erwarten lässt. Die unerwartete Erhöhung der Oxidationskraft von Kupfer(II) erreicht die Natur mit Hilfe einer strukturellen Variation der Ligandsphäre. Diese Anpassung ist so wirksam, dass die oxidierte Form in diesen Enzymen durch anionische Liganden stabilisiert gefunden wird und trotzdem noch ein erheblich stärkeres Oxidationsmittel ist als zum Beispiel die Ammin-Kupfer-Komplexe.
Das in diesem Kapitel zuerst beschriebene Plastocyanin gehört zu einer Klasse von Proteinen mit einheitlichen spektroskopischen Merkmalen. Der Name „blaue Kupferproteine“ weist auf eine charakteristische, ungewöhnlich intensive Absorption bei ca. 16 000 cm−1 hin. Ihre Funktion als 1-Elektronen-Überträger wird durch den in Analogie zu den Ferredoxinen geprägten Sammelnamen „Cupredoxine“ betont. Eine dritte geläufige Bezeichnung für diese Enzymgruppe ist „Typ-I-Kupferproteine“.
Plastocyanin ist ubiquitär in Blaualgen, Algen und allen grünen Pflanzen. Es dient bei der Photosynthese als 1-Elektronentransporter bei hohem Potential. Plastocyanin gehört mit weiteren Cupredoxinen zu einer dritten wesentlichen Klasse löslicher 1-Elektronen-Überträger, die den benötigten Potentialbereich über die Ferredoxine und die Cytochrome hinaus zu hohem Potential hin abschließen. Die elektrochemischen Potentiale von Cupredoxinen erstrecken sich von ca. 0.2 V (Stellacyanin) bis fast zu 0.7 V (Rusticyanin). Für Plastocyanin ist E0' 0.37 V.
Es liegen Strukturanalysen für reduziertes und oxidiertes Plastocyanin verschiedener Pflanzen vor. Abgebildet ist die reduzierte Form des Plastocyanins des Farns Dryopteris crassirhizoma, ein nur 102 Aminosäuren großes Protein (PDB-Code 1KDI, 1.8 Å Auflösung):
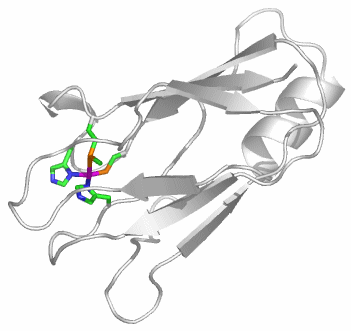
Das aktive Zentrum enthält ein Kupfer(I)-Zentralatom, das von 2 His, 1 Cys und 1 Met koordiniert ist:
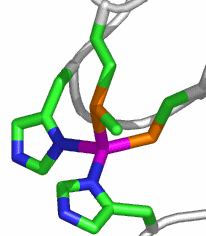
Die Strukturanalyse der oxidierten Form zeigt ein strukturell kaum verändertes aktives Zentrum (PDB-Code 1KDJ, 1.7 Å Auflösung). Die Abstände des Kupferatoms zu den vier Liganden ist nur für die beiden Cu-NHis-Abstände normal; der Cu-SMet-Abstand dagegen ist ungewöhnlich lang, der Cu-SCys-Abstand ist ungewöhnlich kurz (Werte für die oxidierte Form: Cu-NHis 1.93 und 2.07, Cu-SMet 2.92, Cu-SCys 2.26 Å; zum Vergleich die Werte der oxidierten Form von Synechococcus-Plastocyanin, PDB-Code 1BXU, 1.9 Å Auflösung: Cu-NHis 1.97 und 2.01, Cu-SMet 2.94, Cu-SCys 2.14 Å). Die Werte zeigen eine starke Verzerrung des Koordinationspolyeders an, so dass anstelle der auf den ersten Blick vorliegenden tetraedrischen Umgebung besser von einer trigonal-pyramidalen Koordination gesprochen wird mit einer nur geringen Auslenkung des Zentralatoms aus der N2SCys-Ebene.
Die Reorganisationsenergie sollte für ein Kupfer(I,II)-Redoxpaar erheblich sein. Die Vorstellung, dass das Zentralatom in einer Ligandmatrix starr eingebunden ist, löst dieses Problem. Dies ist das Modell vom „entatischen Zustand“ (engl. „entatic state“, „rack-induced“ reactivity), dass sich lange großer Beliebtheit erfreut hat. Bei der quantenchemischen Behandlung des Plastocyanins zeigt sich jedoch, dass bei dieser Vorstellung erhebliche Abstriche zu machen sind (siehe unten).
Der Begriff „blaue Kupferproteine“ weist auf die ungewöhnlich intensive Farbe hin, die eine Folge der starken Absorption (εmax ca. 5000 mol L−1 cm−1) von gelbem Licht ist (Figure 16 in [blue_copper_2004]). Die hohe spezifische Absorption zeigt, dass es sich um eine charge-transfer-Bande handelt. Deren Unterscheidung von den schwächeren Kristallfeldbanden gelingt vor allem durch den Vergleich des UV/Vis-Spektrums mit MCD-Spektren.
Charge-Transfer-Übergänge sind umso intensiver, je stärker die Überlappung von Donor- und Akzeptor-Orbital ist, je höher also der kovalente Charakter der Bindung ist. Die für die Kovalenz maßgebliche Orbital-Wechselwirkung zeigt sich bei der Analyse des elektronischen Grundzustands.
Das Plastocyanin-Zentrum unterscheidet sich durch die ungewöhnlich kurze Cu-SCys-Bindung von normalen Kupferkomplexen. In der oxidierten Form interessiert vor allem die räumliche Lage des spintragenden Orbitals, in das bei der Reduktion ein β-Spin-Elektron eintritt. In den meisten Literaturstellen wird zu dessen Benennung eine Cu-NHis-Bindung, die zweite Cu-NHis-Bindung und die Cu-SMet-Bindung möglichst weitgehend mit der x-, y- und z-Richtung zur Deckung gebracht. In dieser Ausrichtung des Koordinatensystems zeigen quantenchemische Rechnungen das x2−y2-Orbital als magnetisches Orbital an. Das SCys-σ-Donororbital ist nichtbindend in Bezug auf das x2−y2-Orbital. Das senkrecht zur Cu-SCys-Bindung und in der trigonalen Ebene liegende Schwefel-p-Orbital ist jedoch symmetrie-passend für eine Wechselwirkung. Neben einem bindenden MO entsteht ein einfach besetztes antibindendes MO mit lokaler π-Symmetrie, für das die Rechnung und die spektroskopische Charakterisierung eine fast unpolare kovalente Wechselwirkung ergibt. Figure 13 in [blue_copper_2004] zeigt diese Bindungssituation in Form des β-LUMOs. Die Schlussfogerung aus den aktuellen Rechnungen: das Modell vom entatischen Zustand überschätzt den Proteineinfluss – im Plastocyanin-Zentrum ist in der CuN2SCys-Ebene nichts gespannt. Lediglich der Methionin-Ligand, der zu den elektronischen Besonderheiten des Plastocyanins wenig beiträgt, nähert sich bei der Strukturoptimierung in Modellsystemen dem Kupferatom (Figure 5c in [blue_copper_2002]).
Das β-LUMO erscheint im Plastocyanin gegenüber einer normalen Kupfer(II)-Umgebung stabilisiert, wodurch die oxidierende Kraft entsprechend dem höheren E0' steigt. Zum Verständnis reicht das Kristallfeldmodell. Bei einer quantitativen Behandlung zeigt sich, dass die blosse Strukturänderung eines homoleptischen Histidin-Kupfer-Komplexes das Potential weit über den für Plastocyanin gemessenen Wert erhöhen würde. Der Aufbau der zusätzlichen π-Wechselwirkung mit dem anionischen Cysteinato-Liganden senkt nun das zu hohe Potential wieder ein Stück weit – im Einklang mit den bislang abgeleiteten Regeln. Auf die Bedeutung der Kovalenz wird weiter unten eingegangen.
Die binuclearen CuA-Zentren sind hinsichtlich der Proteinsequenz eng mit den Cupredoxinen verwandt. Sie kommen in der Cytochrom-c-Oxidase vor, bei Bakterien werden sie bei der N2O-Reduktase gefunden. Wie die Cupredoxine dienen sie der Weiterleitung eines Elektrons bei hohem Potential. Das Bild zeigt den komplexen Aufbau der N2O-Reduktase von Paracoccus denitrificans, einem denitrifizierenden Bakterium (PDB-Code 1FWX).
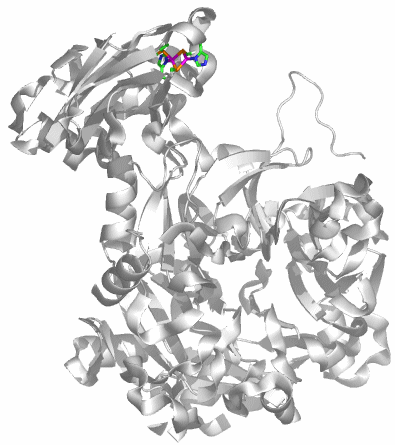
Im aktiven Zentrum fällt vor allem der ungewöhnlich kurze Cu-Cu-Abstand von nur 2.4 Å in der Ruheform mit ihrem mittleren Oxidationsgrad von +1.5 auf. Das Cu2-Paar ist von zwei Cysteinato-Liganden verbrückt, ferner bindet jedes Kupferatom einen terminalen His-Liganden. Weitere Liganden binden deutlich schwächer. Im Bild trägt das linke Cu-Atom in einem größeren Abstand einen Met-Liganden, das rechte Cu-Atom ist in der ausgewählten Kristallstruktur ohne weitere Liganden; andere Strukturbestimmungen an CuA-Zentren zeigen oft eine Koordination des rechten Cu-Atoms durch das O-Atom einer Amidfunktion der Proteinhauptkette:
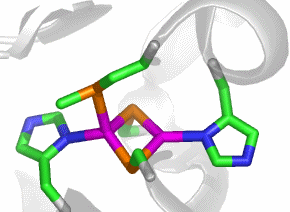
DFT-Rechnungen und spektroskopische Untersuchungen zeigen einen hohen Grad an Kovalenz für die Cu-SCys-σ-Bindungen, ferner kommt eine bindende Cu-Cu-Wechselwirkung hinzu (Figure 1c in [blue_copper_2002]). Das Motiv der Cu-S-Kovalenz wiederholt sich damit bei einem Enzym mit verwandter Funktion.
Eine weitere Gemeinsamkeit zwischen der Plastocyanin- und der CuA-Struktur ist die Y-förmige Koordination dreier nah bindender Liganden. Das x2−y2-Orbital ist in dieser Anordnung deutlich stabilisiert, wodurch die oxidierende Wirkung der zweiwertigen Stufe stark erhöht wird.
Die Elektronenübertragung über eine bestimmte Strecke wird zunehmend erleichtert in der Reihenfolge Vakuum < nichtkovalente Wechselwirkungen (H-Brückenbindungen, ionische Bindungen, polare koordinative Bindungen) < kovalente Bindungen < konjugierte π-Bindungen.
Voraussetzung für den schnellen und gerichteten Elektronentransfer zur Elektronen-Auslass-Position des Plastocyanins, einem mehr als 10 Å entfernten Tyrosin-Rest, ist die Kovalenz der Cu-SCys-Bindung am Beginn der Transportstrecke:
Bei Plastocyanin zeigte sich bereits, dass die Vorstellung, die erzwungene Fixierung eines entatischen Zustands habe eine große Bedeutung für die Funktion eines Kupferenzyms, wohl nicht haltbar ist. Die gefundenen Strukturen stellen wohl Nebenminima in der Metall/Ligand-Energielandschaft dar und keine Positionen besonders hoher Instabilität. Als Folge dieser Erkenntnis ist die Bewertung, welche Koordinationsfigur um Kupfer für welche Oxidationsstufe stabil ist, selbst Gegenstand der aktuellen Diskussion geworden. Die Gleichsetzung Kupfer(II) = JT-gestreckte Oktaeder wie in [Cu(NH3)4(H2O)2]2+ ist derzeit nicht mehr unumstritten. So wird für das Aqua-kupfer(II)-Ion die Zusammensetzung [Cu(H2O)5]2+ ermittelt mit vier fest gebundenen Aqua-Liganden und einem fünften schwach gebundenen Liganden in axialer Position zur besten Ebene durch die vier übrigen Liganden. Die gesamte Struktur ist so irregulär, dass sie die bisherige klare Vorstellung vom „typischen“ Kupfer(II)-Koordinationspolyeder in Frage stellt [cu5aq_2005].
Bei der Klausur … : Zusammenhang zwischen der Energie der Metall-Grenzorbitale und der Reaktivität; Abhängigkeit der Grenzorbitalenergie von der Ligandanordnung.
[blue_copper_2011]
E. I. Solomon, R. G. Hadt:
Recent advances in understanding blue copper proteins.
Coord. Chem. Rev. 2011, 255, 774–789.
doi:
10.1016/j.ccr.2010.12.008
[cu5aq_2005]
P. Frank, M. Benfatto, R. K. Szilagyi, P. D'Angelo, S. D. Longa, K. O. Hodgson:
The Solution Structure of [Cu(aq)]2+ and Its Implications for Rack-Induced Bonding in Blue Copper Protein Active Sites.
Inorg. Chem. 2005, 44, 1922–1933.
doi:
10.1021/ic0400639
[blue_copper_2004]
E. I. Solomon, R. K. Szilagyi, S. DeBeer George, L. Basumallick:
Electronic Structures of Metal Sites in Proteins and Models: Contributions to Function in Blue Copper Proteins.
Chem. Rev. 2004, 104, 419–458.
doi:
10.1021/cr0206317
[blue_copper_2002]
R. K. Szilagyi, E. I. Solomon:
Electronic structure and its relation to function in copper proteins.
Curr. Opin. Chem. Biol. 2002, 6, 250–258.
doi:
10.1016/s1367-5931(02)00304-6
Teilschritte wie
2 CuII + O22− → 2 CuI + O2
oder
CuII + O2•− → CuI + O2
also die Reduktion von Kupfer(II) zur üblicherweise unbeständigen Oxidationsstufe +I durch geläufige Oxidationsmittel sind in der „normalen“ wässrigen Chemie undenkbar. In den Zentren von Hc und CuZnSOD finden diese Reaktionen statt. Die Voraussetzung ist das Zusammentreffen zweier Enzymcharakteristika: (1) die im vorangegangenen Kapitel eingeführte Reaktivitätssteuerung durch die Stabilisierung von Grenzorbitalen und (2) die Steuerung der Elektronenbilanz.
Hämocyanin ist das Sauerstofftransportmolekül von Schnecken und Tintenfischen („Mollusken“) sowie Krebsen, Skorpionen und Spinnen („Arthropoden“). Hc-Einzelmoleküle bilden artspezifisch Oligomere, deren Sinn im Aufbau von Kooperativität des O2-Transports zu liegen scheint (vgl. Mb und Hb). Neben Myoglobin/Hämoglobin und Hämerythrin ist Hämocyanin der dritte O2-Transporter, der in Organismen gefunden wird.
Hämocyanin ist ein Typ-3-Kupferprotein. Charakteristisch für diese Enzymklasse sind zweikernige Kupferzentren, die zur Aktivierung von O2 in der Lage sind – im Sinne von O2-Transport, als Oxygenase oder als Oxidase.
Es liegen Röntgenstrukturanalysen der oxygenierten und der deoxygenierten Form des Hämocyanins von Limulus polyphemus, des Pfeilschwanzkrebses, vor. Der PDB-Eintrag: 1OXY zeigt die Struktur des mit 628 Aminosäuren recht großen Proteins in einer Auflösung von 2.4 Å):
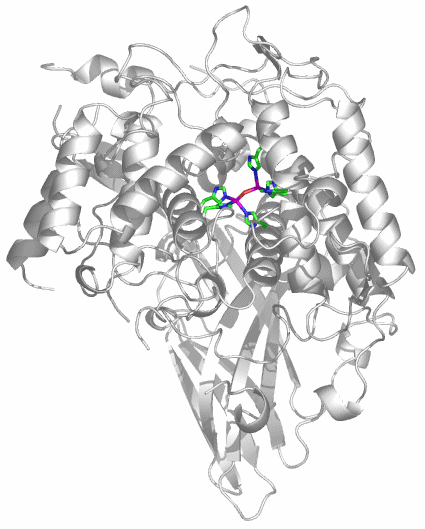
In dem mit Sauerstoff beladenenen aktiven Zentrum beträgt der Abstand der Kupferatome in dem durch einen Peroxido-Liganden verbrückten CuII2-Paar 3.6 Å:

Im deoxygenierten CuI2-Zentrum von Pfeilschwanzkrebs-Hämocyanin (PDB-Eintrag: 1LLA, Auflösung 2.2 Å) ist der Cu-Cu-Abstand mit 4.6 Å deutlich größer:
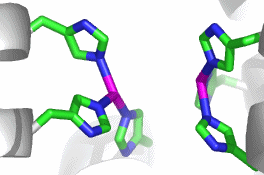
Die Abgabe von Sauerstoff unter Reduktion von Kupfer(II) zu Kupfer(I) steht in krassem Widerspruch zur Laborerfahrung und zur Lage der elektrochemischen Potentiale. Farblose Amminkupfer(I)-Lösungen werden durch Luftsauerstoff schnell und irreversibel zu den bekannten blauen Amminkupfer(II)-Lösungen oxidiert. Dass bei Hc Sauerstoff unter Kupfer(I)-Bildung freigesetzt wird, liegt am Zusammentreffen zweier enzymtypischer Faktoren.
(1) Auch in Hc fehlt eine regulär quadratische Koordination von Liganden, also die Anordnung, die ein stark destabilisiertes x2−y2-Orbital erzeugt und als Folge ein niedriges Potential. Beim Redoxpaar [CuI/II(NH3)4]+/2+ ist genau dies der Fall. Das Potential des Amminkupfer-Paares ist daher mit ca. 0 V kleiner als das Hc-Potential von ca. 0.3–0.4 V.
(2) Ein isoliertes binucleares Zentrum mit den zugänglichen Oxidationszuständen MI2, MIMII und MII2 erlaubt den Umsatz von maximal zwei Elektronen. Die mit höherer Triebkraft verlaufende 4-Elektronen-Reduktion von O2 (E0' = 0.82 V; siehe Anhang II) kann in einem solchen Zentrum nicht ablaufen. Möglich ist bestenfalls der 2-Elektronenprozess Sauerstoff/Peroxid – dessen Potential sich perfekt mit dem Hc-Potential deckt (Anhang II).
Die verwandte und thermodynamisch ebenfalls unmögliche Reaktion
CuII + O2•− → CuI + O2
läuft unter analogen Randbedingungen in Kupfer-Zink-Superoxiddismutase (CuZnSOD) ab:
Auf die Bedeutung der Superoxid-Beseitigung wurde bereits bei der MnSOD eingegangen. Im Vorkommen der einzelnen SODs spiegeln sich die stammesgeschichtlichen Zusammenhänge wider. FeSOD kommt in Prokaryonten vor. MnSOD, die sich wohl aus FeSOD entwickelt hat, kommt ebenfalls in Prokaryonten vor, aber auch in den Mitochondrien höherer Lebewesen. CuZnSOD schließlich – als jüngstes Enzym, in dem zwei Metalle kombiniert sind, die erst in einer schwefelfreien Sauerstoffwelt verfügbar wurden – findet sich im Cytosol aller Eukaryonten. Die Konzentration beträgt hier ca. 10−5 mol L−1. In [cuznsod2] wird errechnet, dass aufgrund der Konzentration und der Aktivität des Enzyms die Lebendauer von Superoxid-Radikalen um den Faktor 1010 gesenkt wird.
CuZnSOD gehört zu den Typ-2-Kupferproteinen, auch „nicht-blaue“ Kupferproteine genannt. Die UV/Vis-Spektren entsprechen im Wesentlichen denjenigen „normal“ koordinierter Kupfer(II)-Komplexe; der starke charge-transfer-Übergang der blauen Kupferproteine fehlt. Die weiter unten behandelte Galactose-Oxidase (GO) gehört ebenfalls zu den Typ-2-Zentren.
Die Struktur humaner CuZnSOD wurde mit einer Auflösung von 1.8 Å bestimmt (PDB-Code 1HL5):
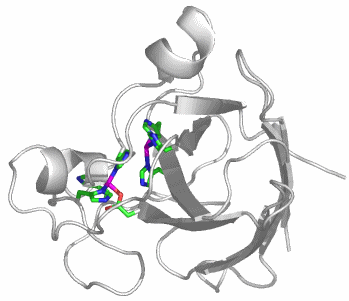
Die Besonderheit des CuZnSOD-Zentrums ist ein zwischen Zink (links) und Kupfer (rechts) verbrückender Histidinato-Ligand. Der Abstand zwischen dem Kupfer(II)-Atom und dem verbrückenden NHis (gestrichelte Linie) beträgt ca. 2 Å in der oxidierten Form und mehr als 3 Å in der reduzierten. Die Strukturanalyse zeigt einen Mittelwert von ca. 2.6 Å, als dessen Ursache die Autoren teilweise Photoreduktion der oxidierten Ruheform während der Röntgenbestrahlung angeben:
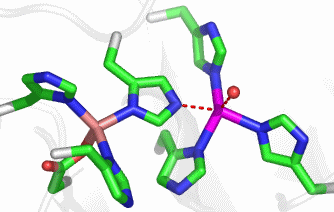
Nach einem Vorschlag in [cuznsod_2005] wird Superoxid auf dem Weg zum aktiven Zentrum protoniert und erreicht das Kupfer-Zentralatom als HO2 (pKA(HO2) = 4.8), wo es an die Stelle des Aqua-Liganden tritt (von anderen Autoren wird üblicherweise mit dem Superoxid-Anion formuliert und die beiden benötigten Protonen werden gesondert zugefügt). Das an Kupfer(II) in der Strukturanalyse gefundene Wassermolekül ist im folgenden Schema nicht berücksichtigt:
Die Nummerierung gilt für das in [cuznsod_2003] beschriebene humane Enzym. Oft untersucht ist Rinder-Erythrozyten-CuZnSOD, in der Kupfer von den His-Resten 44, 46, 61 und 118 koordiniert ist.
Der Katalysecyclus betont die Aussage, dass die Elektronendifferenz der verfügbaren Oxidationszustände die ablaufende Reaktion determiniert. Da Zink redox-inaktiv ist – seine Bedeutung wird in der Erhöhung der Acidität des verbrückenden His gesehen – sind ausschließlich 1-Elektronenschritte möglich. Genau dies verlangt die Superoxid-Disproportionierung.
Auch das elektrochemische Potential liegt im notwendigen Bereich. Zur Erinnerung: während das Potential eines Sauerstoff-Transporters dem Potential des genutzten Redoxprozesses entsprechen muss, so muss das Potential eines Disproportionierungskatalysators zwischen den Potentialen der beiden katalysierten Einzelschritte liegen.
Mit CuZnSOD sind nun die meisten SODs beschrieben worden (es gibt noch eine NiSOD). Ein vergleichender Übersichtsartikel ist [sods_2010].
[sods_2010]
I. A. Abreu, D. E. Cabelli:
Superoxide dismutases—a review of the metal-associated mechanistic variations.
Biochim. Biophys. Acta 2010, 1804, 263–274.
doi:
10.1016/j.bbapap.2009.11.005
[cuznsod_2005]
V. Pelmenschikov, P. E. M. Siegbahn:
Copper-Zinc Superoxide Dismutase: Theoretical Insights into the Catalytic Mechanism.
Inorg. Chem. 2005, 44, 3311–3320.
doi:
10.1021/ic050018g
[cuznsod_2003]
R. W. Strange, S. Antonyuk, M. A. Hough, P. A. Doucette, J. A. Rodriguez, P. J. Hart, L. J. Hayward, J. S. Valentine, S. S. Hasnain:
The Structure of Holo and Metal-deficient Wild-type Human Cu, Zn Superoxide Dismutase and its Relevance to Familial Amyotrophic Lateral Sclerosis.
J. Mol. Biol. 2003, 328, 877–891.
doi:
10.1016/S0022-2836(03)00355-3
Die Chemie von Hämocyanin und CuZnSOD ist durch die Festlegung der Elektronenbilanz aufgrund der Zahl der Redoxzentren und deren verfügbarer Oxidationsstufen festgelegt. Die Katalyse einer 2-Elektronen-Oxidation durch ein einkerniges Kupferprotein muss daher irritieren. Ist in Galactose-Oxidase Kupfer(III) in Betracht zu ziehen?
Die von Pilzen ausgeschiedene Galactose-Oxidase (GO) oxidiert in einem 2-Elektronenschritt primäre Alkohole zu Aldehyden:
RCH2OH + O2 = RCHO + H2O2
Andere oxidierbare funktionelle Gruppen werden nicht angegriffen. So wird im natürlichen Substrat d-Galactose die Hydroxylfunktion an C6 oxidiert, ohne dass die Aldehydfunktion an C1 oder die sekundären Hydroxylgruppen angegriffen würden:
d-Galactose wird selektiv zu Galacto-1,6-dialdose oxidiert. Der Sinn der Reaktion ist unklar, da keine weitere Verwendung von Galactodialdose bekannt ist. Eine Vermutung ist die, dass GO-ausscheidende Pilze das in die Umgebung entlassene H2O2 zu ihrem Schutz freisetzen.
Für GO liegt eine Kristallstrukturbestimmung in einer Auflösung von 2.3 Å vor (PDB-Code 1T2X). Das Apoprotein ist auffallend reich an β-Faltblattabschnitten:
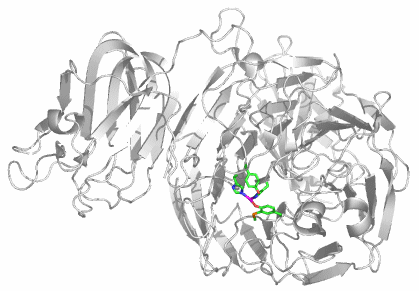
Das aktive Zentrum enthält ein Kupfer-Zentralatom, das im üblichen Abstand von ca. 2 Å für die erste Koordinationssphäre nur drei Ligandatome bindet. Zwei His-Liganden werden dabei durch ein in ungewöhnlicher Weise modifiziertes Tyr ergänzt: durch Substitution eines ortho-Wasserstoffatoms durch das Schwefelatom eines benachbarten Cys-Restes wird der Tyr-Ligand in einer Konformation fixiert, bei welcher der aromatische Ring senkrecht zur CuN2O-Ebene steht:
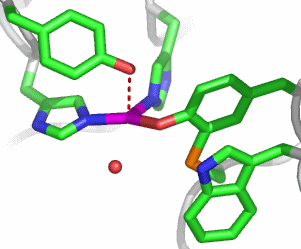
Die im Bild von links oben hereinragende zweite Tyr-Seitenkette befindet sich in einem Abstand von 2.73 Å vom Kupferatom. Die Wechselwirkung ist schwächer als beim fester gebundenen Tyr. Die naheliegende Frage, ob der Cu-Tyr-Abstand noch ausreicht, die Deprotonierung der phenolische Hydroxylgruppe zu unterstützen, wird durch neuere Untersuchungen gegenstandslos: das entferntere Tyr trägt an O kein H-Atom, es liegt in der oxidierten Enzymform vielmehr als Tyr•-Radikal-Ligand vor.
Scheme 1 (Cyclus) und Fig. 4 (Struktur des Übergangszustands der H-Übertragung und Struktur des Ketylradikalkomplexes) in [go_2003] zeigen die derzeitigen Vorstellungen vom Ablauf der Katalyse. Die H-Atom-Übertragung verlangt die Vertikalstellung von Tyr272. Dies trifft auch in der abweichenden Formulierung des Katalysecyclus in Fig. 4 in [go_2004] zu. Eine Übersicht über die derzeitigen Vorstellungen gibt [go_2005].
CcO ist das letzte Enzym der Atmungskette. CcO wird durch vier Äquivalente Cytochrom c reduziert, um anschließend ein Sauerstoffmolekül in einem Schritt zu Wasser zu reduzieren. Die gewonnene Energie wird als Protonengradient gespeichert. Die große Zahl von vier Elektronen für die Reaktion
O2 + 4 e− + 4 H+ = 2 H2O
stellt besondere Anforderungen an den Aufbau des aktiven Zentrums.
CcO besteht aus 26 Proteinketten mit insgesamt 3614 Aminosäuren. Es liegt eine Strukturanalyse von Rinderherz-CcO im oxidierten Zustand mit 1.8 Å Auflösung vor (PDB-Code 1V54). Das O2-reduzierende Häm-a3-CuB-Zentrum kommt zusammen mit einem weiteren Häm-a in zwei Proteinketten vor. Abgebildet ist eine dieser beiden fast ausschließlich aus α-Helices bestehenden Ketten (A), zusätzlich ist im oberen Teil der Abbildung die an Faltblattabschnitten reichere Kette B. Zwischen A und B findet sich ein elektronenleitendes CuA-Zentrum:
Das abgebildete Protein reicht durch die innere Mitochondrienmembran. Der untere Teil ragt in das Innere der Organelle, der obere Teil ragt in den Transmembranraum zwischen innerer und äußerer Mitochondrienmembran. Die durch Cyt c herantransportierten Elektronen werden vom CuA-Zentrum angenommen und an das Häm-a-Zentrum weitergegeben. Der Elektronenfluss endet beim sauerstoffreduzierenden Häm-a3-CuB-Zentrum. Die vier Protonen der Bruttogleichung stammen aus dem Inneren des Mitochondriums (im Bild unten).
Beim Häm-a3-CuB-Zentrum fällt vor allem die Umgebung des Kupferatoms auf. Diese besteht aus drei His-Liganden, deren einer eine direkte Bindung zu einem Tyr-Rest aufweist:
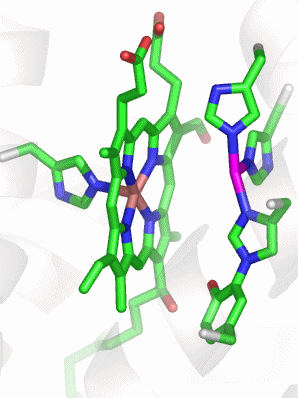
Vgl. den Ablauf in Fig. 2 in [cco_2005] und in Fig. 2 in [cco_2004].
Die Kopplung des Protonentransfers an den Redoxprozess ist Gegenstand der aktuellen Diskussion [cco_2003], [cco_2004].
Bei der Klausur … : Koordinationsgeometrie bei verschiedenen dn-Konfigurationen; formale und physikalische (spektroskopische) Oxidationsstufe; Lage eines spintragenden Orbitals im Kristallfeld.
[go_2005]
J. W. Whittaker:
The radical chemistry of galactose oxidase.
Arch. Biochem. Biophys. 2005, 433, 227–239.
doi:
10.1016/j.abb.2004.08.034
[go_2003]
T. Lovell, F. Himo, W.-G. Han, L. Noodleman:
Density functional methods applied to metalloenzymes.
Coord. Chem. Rev. 2003, 238–239, 211–232.
doi:
10.1016/S0010-8545(02)00331-4
[go_2004]
S. G. Minasian, M. M. Whittaker, J. W. Whittaker:
Stereoselective Hydrogen Abstraction by Galactose Oxidase.
Biochemistry 2004, 43, 13683–13693.
doi:
10.1021/bi048554s
[cco_2005]
M. Brunori, A. Giuffrè, P. Sarti:
Cytochrome c oxidase, ligands and electrons.
J. Inorg. Biochem. 2005, 99, 324–336.
doi:
10.1016/j.jinorgbio.2004.10.011
[cco_2004]
A. Namslauer, P. Brzezinski:
Structural elements involved in electron-coupled proton transfer in cytochrome c oxidase.
FEBS Letters 2004, 567, 103–110.
doi:
10.1016/j.febslet.2004.04.027
[cco_2003]
T. Tsukihara, K. Shimokata, Y. Katayama, H. Shimada, K. Muramoto, H. Aoyama, M. Mochizuki, K. Shinzawa-Itoh, E. Yamashita, M. Yao, Y. Ishimura†, S. Yoshikawa:
The low-spin heme of cytochrome c oxidase as the driving element of the proton-pumping process.
PNAS 2003, 100, 15304–15309.
doi:
10.1073/pnas.2635097100
Neben der Nitrogenase ist das Photosystem II (PSII) der Dauerbrenner bioanorganischer Forschung. Nachdem Kok 1970 die Grundlage für ein 5-Zustände-Modell gelegt hatte, blieb und bleibt die Struktur des sauerstofffreisetzenden Komplexes („OEC“ von oxygen-evolving complex) der Schlüssel zum Verständnis der Funktion. Obwohl die Kenntnis der Struktur in den vergangenen Jahren durch Einkristallstrukturanalyse erheblich zugenommen hat, bleiben viele Fragen, da die derzeit beste Auflösung lediglich 3 Å beträgt, und da es große Unsicherheiten gibt, inwieweit Strahlenschäden beim Röntgenexperiment das Ergebnis beeinflussen.
Die Wasseroxidation durch Photosynthese, bei der gemäß
2 H2O → O2 + 4 H+ + 4 e−
Sauerstoff, Protonen und vier Reduktionsäquivalente durch eine lichtgetriebene Reaktion entstehen, findet in der Thylakoid-Membran der Chloroplasten aller grünen Pflanzen statt [oec_2004]. Bereits Anfang der 1970er Jahre publizierte Kok ein Blitzlicht-Experiment, das zur Formulierung eines Fünf-Zustandsmodells führte. Dieses zunächst ohne Kenntnis molekularer Details aufgestellte, phänomenologische Modell unterteilte die Wasseroxidation in Einzelschritte: ausgehend von einem maximal reduzierten Zustand S0 führt die Absorption des ersten Lichtquants durch 1-Elektronen-Oxidation des aktiven Zentrums zu S1. Dieser Vorgang wiederholt sich noch dreimal, wobei S2, S3 und S4 entstehen. S4 stellt den höchstoxidierten Zustand dar, der Sauerstoff freisetzt und dabei in den um vier Elektronen reicheren S0-Zustand zurückkehrt. Die einzelnen Elektronen, die beim Durchlaufen der S-Zustände entzogen werden, reduzieren ein räumlich benachbartes Tyr-Radikal, das durch Reduktion des durch Photooxidation entstandenen radikalischen P680+-Zustands entstanden war. Das nach Anregung des P680-Zentrums freigesetzte Elektron wird durch Plastochinon auf Photosystem I übertragen, wo durch weitere Lichtenergie die für den Calvin-Cyclus benötigten, stark reduzierenden Niedrig-Potential-Elektronen in Form von NADPH erzeugt werden.
Das Kok-Experiment nach V. P. Shinkarev, Biophys. J. 2005, 88, 412–421. Auf der Ordinate ist die von dunkeladaptierten Spinatchloroplasten nach einem Blitz der angegebenen Nummer freigesetzte Sauerstoffmenge aufgetragen. Welches ist der Ruhezustand eines Chloroplasten?
Die mit 3 Å bestaufgelöste Struktur ist in 2AXT hinterlegt und wird in [oec_2007] beschrieben. Eine Übersicht über die derzeit vorliegenden Strukturanalysen gibt [oec_2008]. Die Beugungsdaten werden durch röntgenspektroskopische Untersuchungen ergänzt [oec_2008a]. Die Strukturanalyse zeigt einen großen Transmembran-Protein-Cofaktor-Komplex, der die Thylakoidmembran eines Chloroplasten durchdringt. Der Komplex hat C2-Symmetrie und enthält in jeder Untereinheit einen OEC (gelbe Kreise):
Die asymmetrische Einheit ist aus 18 Proteinketten aufgebaut, die von einer Fülle licht- und elektronenleitender Cofaktoren durchzogen ist, vor allem von Chlorophyllmolekülen. Die hohe Elektronendichte des OEC wird als CaMn4-Kern interpretiert, der im folgenden Bild etwas unterhalb der Mitte zu sehen ist (Mn violett, Ca grün).
Der hohe Anteil an Cofaktoren ist in den beiden folgenden Bildern hervorgehoben, die in derselben Orientierung wie die Totalansicht zuerst nur den Proteinanteil einschließlich des OEC zeigen …
… und anschließend nur die Cofaktoren, wieder einschließlich des OEC:
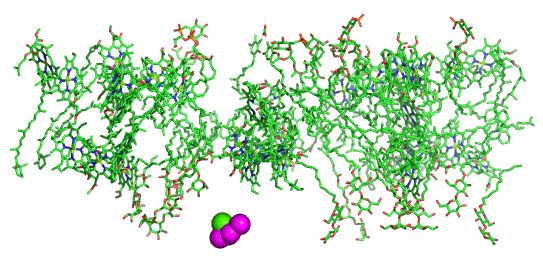
Die Interpretation der Elektronendichte des OEC lässt viele Fragen offen. Die Autoren von 2AXT ordnen keine nichtproteinogenen Liganden zu, so dass viele Koordinationsstellen frei bleiben:
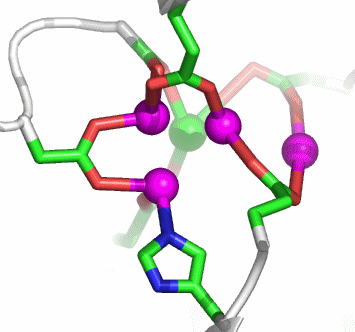
Im Einklang mit den durchweg hohen Oxidationsstufen der Manganatome wird der OEC vor allem von anionischen Carboxylato-Liganden gehalten. Neben den üblichen Asp- und Glu-Seitenketten ist hierunter auch die Carboxylatgruppe eines C-terminalen Alaninrestes (im Bild oben rechts). Die Abstände der Manganatome sind dieselben, wie sie bei Modellkomplexen und bei Manganoxiden in kantenverknüpften Mn(μ-O)2Mn-Fragmenten gefunden werden, weshalb bei den Strukturvorschlägen der meisten Autoren dieses Strukturfragment allgegenwärtig ist. Das folgende Schema zeigt eines der Beispiele:
Derzeit werden als Oxidationszustände diskutiert:
S0: II,III,IV,IV oder III,III,III,IV,
S1: III,III,IV,IV,
S2: III,IV,IV,IV,
S3: IV,IV,IV,IV.
Eine Beschreibung für S4 ist unklar. Autoren, die eine O-O-Bindung erst im Schritt S4→S0 formulieren, sehen S4 als MnIV4(O•−). Alternativ wird die Bildung einer ersten O-O-Bindung nach Erreichen von S3 diskutiert, wobei dem Mangancluster vorzeitig zwei Oxidationsäquivalente entzogen werden.
Einigkeit herrscht über die Kopplung des Elektronenentzugs mit Deprotonierungsschritten bei jeder Photonenaufnahme [oec_2007a]. Schematisch:
Die Elektron-Proton-Kopplung erklärt den nur allmählichen Potentialanstieg des OEC durch die zunehmende Stabilisierung der ansteigenden Oxidationsstufen durch immer mehr anionische Liganden. Zugleich schafft diese Kopplung mehrere Brønsted-basische Zentren, die beim S4→S0-Schritt Protonen der zu oxidierenden Wassermoleküle aufnehmen können.
Wir könnten mit Kenntnis der Cytochrom-c-Oxidase fordern, dass die Natur es vielleicht eher vermeiden wird, Peroxid zu binden während auf das nächste Photon gewartet wird. Damit würden dann Vorstellungen übrig bleiben, nach denen zuerst vier Oxidationsäquivalente akkumuliert werden und erst anschließend, im S4→S0-Schritt, die O-O-Verknüpfung stattfindet. Schematisch:
Der Aqua-Ligand ist im Schema bewusst unklar koordiniert, da umstritten ist, ob er ein Ligand an Calcium oder an einem der Manganatome ist. Die beiden Protonen werden von Oxido-Liganden gebunden, die durch die Reduktion der Metallzentren basischer werden. All diese vagen Aussagen hängen nicht nur mit den experimentellen Schwierigkeiten rund um das Photosystem II zusammen (Präparation reiner, definierter S-Zustände), sondern auch damit, dass die computerchemische Modellierung dieses komplexen Systems noch in den Kinderschuhen steckt [oec_2008b] und [oec_2009].
Das aktive Zentrum im OEC weist Strukturmerkmale einfacher Mangan-(hydr)oxide auf. Das gleichzeitige Auftreten von Kanten- und Eckenverknüpfung von MnO6-Oktaedern tritt zum Beispiel bei Pyrolusit auf, einer kristallinen MnO2-Modifikation mit Rutil-Struktur; Abbildungen hierzu sind im Abschnitt „Eisen(III)-(hydr(oxide)“ der Vorlesung „Biomineralisation“ zusammengestellt.
Die Modellierung des OEC des Photosystems II ist ein Schwerpunkt bioanorganischer Forschung. Die Arbeiten sind, ähnlich wie bei den im Folgenden beschriebenen Versuche zur Hydrogenase, durch die Hoffnung auf die Entwicklung wirksamer Katalysatoren gerichtet. Das Ziel ist nicht allein den Nachbau des Photosystems, sondern die Entwicklung wirksamerer, technisch nutzbarer Katalysatoren (der Wirkungsgrad der Photosynthese ist nicht so hoch, als dass der Einsatz grüner Pflanzen zu diesem Zweck attraktiv wäre). Einen Überblick über den Stand der Forschung gibt [oec_2009a].
Die neuen Publ. demnächst einbauen:
[oec_review_2011]
M. M. Najafpour, Govindjee:
Oxygen evolving complex in Photosystem II: Better than excellent.
Dalton Trans. 2011, 40, 9076-9084.
doi:
10.1039/C1DT10746A
[oec_2011]
Y. Umena, K. Kawakami, J.-R. Shen, N. Kamiya:
Crystal structure of oxygen-evolving photosystem II at a resolution of 1.9 Å.
Nature 2011, 473, 55–60.
doi:
10.1038/nature09913
[oec_2009]
P. E. M. Siegbahn:
Structures and Energetics for O2 Formation in Photosystem II.
Acc. Chem. Res. 2009, 42, 1871–1880.
doi:
10.1021/ar900117k
[oec_2009a]
X. Sala, I. Romero, M. Rodriguez, L. Escriche, A. Llobet:
Molekulare Katalysatoren für die Oxidation von Wasser zu Disauerstoff.
Angew. Chem. 2009, 121, 2882–2893.
doi:
10.1002/ange.200802659
[oec_2008]
J. Barber:
Crystal Structure of the Oxygen-Evolving Complex of Photosystem II.
Inorg. Chem. 2008, 47, 1700–1710.
doi:
10.1021/ic701835r
[oec_2008a]
J. Yano, V. K. Yachandra:
Where Water Is Oxidized to Dioxygen: Structure of the Photosynthetic Mn4Ca Cluster from X-ray Spectroscopy.
Inorg. Chem. 2008, 47, 1711–1726.
doi:
10.1021/ic7016837
[oec_2008b]
E. M. Sproviero, J. A. Gascon, J. P. McEvoy, G. W. Brudvig, V. S. Batista:
Computational studies of the O2-evolving complex of photosystem II and biomimetic oxomanganese complexes.
Coord. Chem. Rev. 2008, 252, 395–415.
doi:
10.1016/j.ccr.2007.09.006
[oec_2007]
J. Kern, J. Biesiadka, B. Loll, W. Saenger, A. Zouni:
Structure of the Mn4–Ca cluster as derived from X-ray diffraction.
Photosynth. Res. 2007, 92, 389–405.
doi:
10.1007/s11120-007-9173-1
[oec_2007a]
T. J. Meyer, M. H. V. Huynh, H. H. Thorp:
Zur möglichen Rolle des protonengekoppelten Elektronentransfers (PCET) bei der Oxidation von Wasser durch das Photosystem II.
Angew. Chem. 2007, 119, 5378–5399.
doi:
10.1002/ange.200600917
[oec_2004]
N. Nelson, A. Ben-Shem:
The complex architecture of oxygenic photosynthesis.
Nature Rev. Mol. Cell Biol. 2004, 5, 1–12.
doi:
10.1038/nrm1525
Bei den Hydrogenasen – wasserstoffspaltenden und -bildenden Enzymen – werden Metalloenzyme von metallfreien Hydrogenasen unterschieden. Die Metalloenzyme, die in [NiFe]-, [FeFe]- und [Fe]-Hydrogenasen zerfallen, nehmen aufgrund von Carbonyl- und Cyanido-Liganden an Eisen eine Sonderstellung ein. Es stellt sich die Frage nach dem Nutzen dieser scheinbar „unbiologischen“ Liganden und ihrer Biosynthese. Die Aktivität von Hydrogenasen ist hoch: eine mit NiFe-Hydrogenase belegte Elektrode setzt so schnell Wasserstoff um als wäre sie mit Platin belegt. Gelänge die Übertragung sauerstoffbeständiger Hydrogenasen in die Technik, könnten Brennstoffzellen gebaut werden, in denen die Elektrodenräume nicht mehr durch Membranen getrennt werden müssten.
Hydrogenasen erlauben es Anaerobiern zum einen, Protonen als terminale Elektronenacceptoren zu nutzen, zum anderen können H2-verbrauchende Organismen Wasserstoff als Reduktionsäquivalent einsetzen. In beiden Fällen katalysieren Hydrogenasen die Reaktion:
H2 ⇌ 2 H+ + 2 e−
NiFe-Hydrogenasen, die bestuntersuchten Hydrogenasen, katalysieren die heterolytische Spaltung von H2 und die anschließende Oxidation des Hydrids und fördern so die Hinreaktion [h2ases_2002].
Es liegt eine Serie hochaufgelöster Strukturbestimmungen von Nickel-Eisen-Hydrogenase von Desulfovibrio vulgaris vor, bei welchen sich die Vorbehandlung der Kristalle unterscheiden. Dargestellt ist die mit einer Auflösung von 1.35 Å gelöste Struktur, die wahrscheinlich das reaktionsbereite reduzierte Enzym zeigt: der Kristall wurde zuerst mit H2, dann mit CO behandelt, anschließend unter Bedingungen photolysiert, welche die Abspaltung des vorher zugefügten CO sicherstellen sollten, und anschließend wieder mit H2 behandelt (PDB-Code 1UBU). Das Holoenzym besteht aus zwei Proteinketten (transparent hellgrau und dunkler grau dargestellt), die insgesamt vier eisenhaltige Reaktionszentren einschließen: ein tief im Inneren lokalisiertes Nickel-Eisen-Zentrum sowie drei Eisen-Schwefel-Cluster, die das verborgene aktive Zentrum mit der Enzymoberfläche „verdrahten“:
Im folgenden Ausschnitt liegt das aktive Zentrum oben, darunter stellen drei Ferredoxine den Elektronenfluss von und zum aktiven Zentrum sicher. Nur eines der Ferredoxine – das obere – hat die geläufige [4Fe-4S]-Struktur, dem darunter liegenden fehlt ein Eisenatom. Solche [3Fe-4S]-Zentren dienen demselben Zweck wie die übrigen Fds. Unten schließlich ein vierkerniges Fd, das eine ungewöhnliche Umgebung aufweist, indem einer der üblichen Cys-Liganden durch His ersetzt ist:
Das aktive Zentrum hat eine völlig unerwartete Struktur: ein biologisch aktiver Zweikernkomplex, der kaum den Erwartungen an ein Biomolekül entspricht:
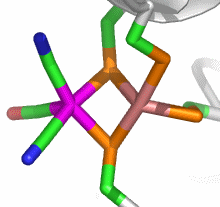
Das Eisenatom (links) ist von Cyanido- und Carbonylliganden koordiniert. Der Ni-Fe-Abstand beträgt 2.61 Å. Die Koordinationslücke im aktiven Zentrum lässt sich leicht sichtbar machen: Behandlung von NiFe-Hydrogenase mit CO führt zur reversiblen Blockierung des Enzyms. Die Strukturanalyse (PDB-Code 1UBO) zeigt die Bindung eines schwach gewinkelten Carbonyl-Liganden am Nickelatom:
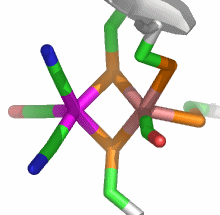
In der Literatur herrscht Einigkeit über die heterolytische Spaltung von H2 (Übersicht: [h2ases_2007]). Das dabei gebildete Hydrid tritt wohl als Hydrido-Brücke zwischen die beiden Metall-Atome, während ein Proton von Cys546 übertragen und von diesem weitergegeben wird. Man beachte, dass bei der derzeitigen Auflösung der Strukturanalysen keine Wasserstofflagen experimentell bestimmt werden können. Es konnte jedoch durch spezielle NMR-Techniken abgesichert werden, dass ein Hydrido-Ligand vorliegt [h2ases_2003]. Der dritte relativ einheitlich formulierte Punkt betrifft die Oxidationsstufen des Nickelatoms, das im Verlauf des Cyclus zwischen den Stufen +II und +III wechselt. Diese Einzelheiten sind in dem abgebildeten Minimalcyclus berücksichtigt.
Hydrido-Liganden sind – auch in einer wässrigen Umgebung – ausreichend stabilisiert, wenn die Metall-H-Bindung hinreichend kovalent ist. Ein high-spin-Eisen(II)-Zentrum würde diese Forderung sicher nicht erfüllen. Das Eisen(II)-Atom liegt jedoch – wie auch das Nickel-Atom – im low-spin-Zustand vor. Dies bedingt ein auf den μ-H-Donorliganden gerichtetes leeres Akzeptororbital (das im high-spin-Fall halbbesetzt wäre). Cyanido- und Carbonyl-Liganden sind unumgänglich, da es der wirksamsten Starkfeld-Liganden bedarf, um trotz der Anwesenheit der nur schwach aufspaltenden, π-basischen Cysteinato-Liganden in der Summe zu einer low-spin-stabilisierenden Kristallfeldaufspaltung zu gelangen.
Der Cyanido-Ligand scheint aus der C-N-Funktion von Carbamoylphosphat zu stammen.
Nach schrittweisem Aufbau der C-N-Dreifachfachbindung (Fig. 3 in [h2ases_2003a]) erfolgt die Übertragung des CN-Fragments durch einen reduktiven Reaktionsschritt von einer Cys-S-CN-Vorstufe auf Eisen. Die Bildung von freiem CN− wird auf diese kontrollierte Weise umgangen.
Eine Strukturanalyse am Desulfovibrio-desulfuricans-Enzym liegt in einer Auflösung von 1.6 Å vor (PDB-Code: 1HFE). Das Holoenzym enthält zwei [4Fe-4S]-Cluster, die den Weg der ausgetauschten Elektronen von der Proteinperipherie in das aktive Zentrum nachzeichnen, und einen sechskernigen Eisencluster. Das dargestellte Protein ist in ungewöhnlicher Weise von einer zweiten ringförmigen Proteinkette wie mit einem Gürtel umgeben. Da dieses Gürtelprotein keine unmittelbaren Kontakte zum anorganischen Teil hat, ist es der Übersichtlichkeit halber weggelassen:
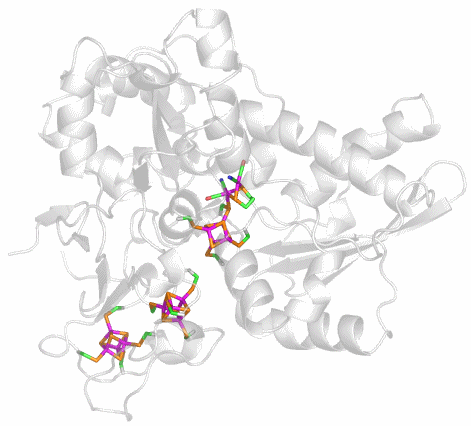
Das aktive Zentrum, der sechskernige Eisencluster, hat einen ebenso „unbiologisch“ anmutenden Aufbau wie das aktive Zentrum der [NiFe]-Hydrogenase. Da es ohne Vorbild ist, gelang es bei den ersten Strukturanalysen nicht, die Liganden mit ausreichender Sicherheit zuzuordnen. Eine aktuelle, DFT-untertützte Strukturanalyse am Clostridium-pasteurianum-Enzym in einer Auflösung von 1.4 Å (PDB-Code: 3C8Y) hat hier weitgehende Klarheit geschaffen.
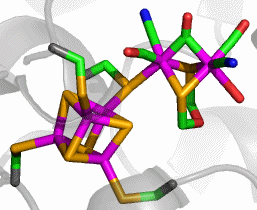
An einen [4Fe-4S]-Cluster angeknüpft, fällt vor allem eine binucleare Einheit auf, in der zwei Eisenatome in eigenartiger Weise koordiniert sind: neben je einem Carbonyl- und einem Cyanido-Liganden werden die beiden Metallatome durch einen bifunktionellen Liganden verbrückt, der als Dithiomethylether angesehen wird. Desweiteren tritt ein μ2-CO-Ligand auf, am rechten Eisenatom schließlich ein Aqua/Hydroxido-Ligand. Auffallend ist der kurze Eisen-Eisen-Abstand von 2.55 Å, der eine Metall-Bindung als möglich erscheinen lässt [h2ases_2005]. Die Strukturanalyse entspricht der IR-spektroskopischen Analyse (Figure 3 in [h2ases_2010]).
Um über eine Metall-Metall-Bindung zu entscheiden, müssten die Oxidationsstufen der Eisenatome bekannt sein. Der derzeitige Kenntnisstand zu dieser Frage ist in [h2ases_2007] dargestellt. Es ist möglich, dass reduzierte [FeFe]-Hydrogenase die bislang in der Biologie nicht gefundene Oxidationsstufe +I an Eisen aufweist, die in synthetischen Modellverbindungen seit langem etabliert ist.
Die aktuellen Vorstellungen zum Katalysecyclus zeigen Ähnlichkeit mit dem Ablauf beim Nickel-Eisen-Enzym. Sicher scheint auch hier die Heterolyse der H-H-Bindung (Figure 24 in [h2ases_2007]).
Bei Archaeae wird eine einkernige Eisenhydrogenase gefunden, welche die Umwandlung eines H2-Moleküls durch Hydridübertragung auf einen Methylentetrahydromethanopterin-Cofaktor in ein 2e-Reduktionsäquivalent katalysiert. In der Literatur ist neben der Bezeichnung [Fe]-Hydrogenase auch „Hmd“ (von H2-forming methylenetetrahydromethanopterindehydrogenase) üblich. Die katalysierte Reaktion ist:
H2 + R2N-CH=N+R2 → H+ + R2N-CH2-NR2
Der reduzierte Cofaktor dient anschließend der CO2-Reduktion. Eine Strukturanalyse mit 1.75 Å Auflösung zeigt eine ungewöhnlichen Ligandausstattung am Eisenzentrum (3F47).
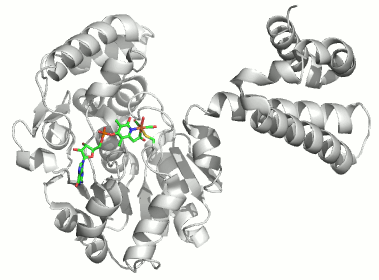
Die Umgebung des Eisenatoms, dessen Oxidationsstufe +II sein könnte, ist mit zwei Carbonyl- und einem 2-Acylpyridin-Chelatligand wieder auf low-spin ausgerichtet (der sechste Ligand, im Bild als O-Atom dargestellt, ist nicht eindeutig identifiziert).
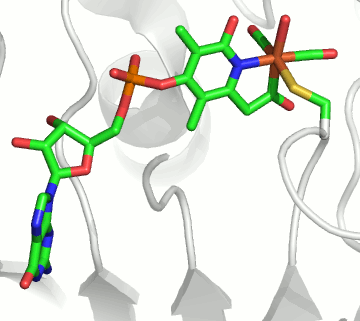
Der Guanylylpyridon-Ligand wird in [h2ases_2008] in der Form des Pyridinol-Tautomers gesehen (Guo = Guanosin):
Der Acyl-Ligand ist für ein Enzym ungewöhnlich, in der Organometallchmie aber wohlbekannt. Die Untersuchung der Carbonyl-Liganden einschließlich der Bindung eines dritten CO- oder eines Cyanido-Liganden ist ein sehr schönes Beispiel für den Einsatz der IR-Spektroskopie in der Charakterisierung eines Metalloenzyms [feh2ase_ir_2004].
Aktuelle Vorschläge zum Katalysecyclus greifen das Grundprinzip auf, dass ein H2-Molekül bei der Koordination an das Zentralmetall polarisiert und dann heterolytisch gespalten wird, worauf das Hydrid-Ion auf das Substrat übertragen wird (Scheme 12 in [feh2ase_2011]).
Die zunehmende Kenntnis zu Hydrogenasen lädt zu Verallgemeinerungen ein. So finden Sie Ideen zur Bedeutung gemeinsamer Strukturprinzipien in [h2ases_2010a], wo die Acyl-CO-Funktion mit dem Brücken-CO-Ligand in [FeFe]-Hydrogenase gleichgesetzt wird. Alle nur denkbaren Analogien zwischen Hydrogenase-Katalyse und organometallchemischen Regeln werden in [h2ases_organomet_2010] ausgeleuchtet.
Bei der Klausur … : Stellung von π-Säuren in der spektrochemischen Reihe; Darstellung der π-Säure–Zentralmetall-Wechselwirkung im Orbitalbild.
[feh2ase_2011]
M. J. Corr, J. A. Murphy:
Evolution in the understanding of [Fe]-hydrogenase.
Chem. Soc. Rev. 2011, 40, 2279–2292.
doi:
10.1039/C0CS00150C
[h2ases_organomet_2010]
J. C. Gordon, G. J. Kubas:
Perspectives on How Nature Employs the Principles of Organometallic Chemistry in Dihydrogen Activation in Hydrogenases.
Organometallics 2010, 29, 4682–4701.
doi:
10.1021/om100436c
[h2ases_2010]
E. M. Shepard, S. E. McGlynn, A. L. Bueling, C. S. Grady-Smith, S. J. George, M. A. Winslow, S. P. Cramer, J. W. Peters, J. B. Broderick:
Synthesis of the 2Fe subcluster of the [FeFe]-hydrogenase H cluster on the HydF scaffold.
PNAS 2010, 107, 10448–10453.
doi:
10.1073/pnas.1001937107
[h2ases_2010a]
M. T. Stiebritz, M. Reiher:
A Unifying Structural and Electronic Concept for Hmd and [FeFe] Hydrogenase Active Sites.
Inorg. Chem. 2010, 49, 5818–5823.
doi:
10.1021/ic902529c
[h2ases_2007]
P. E. M. Siegbahn, J. W. Tye, M. B. Hall:
Computational Studies of [NiFe] and [FeFe] Hydrogenases.
Chem. Rev. 2007, 107, 4414–4435.
doi:
10.1021/cr050185y
[h2ases_2005]
X. Liu, S. K. Ibrahim, C. Tard, C. J. Pickett:
Iron-only hydrogenase: Synthetic, structural and reactivity studies of model compounds.
Coord. Chem. Rev. 2005, 249, 1641–1652.
doi:
10.1016/j.ccr.2005.04.009
[feh2ase_ir_2004]
E. J. Lyon, S. Shima, R. Boecher, R. K. Thauer, F.-W. Grevels, E. Bill, W. Roseboom, S. P. J. Albracht:
Carbon Monoxide as an Intrinsic Ligand to Iron in the Active Site of the Iron?Sulfur-Cluster-Free Hydrogenase H2-Forming Methylenetetrahydromethanopterin Dehydrogenase As Revealed by Infrared Spectroscopy.
J. Am. Chem. Soc. 2004, 126, 14239–14248.
doi:
10.1021/ja046818s
[h2ases_2003]
M. Brecht, M. van Gastel, T. Buhrke, B. Friedrich, W. Lubitz:
Direct Detection of a Hydrogen Ligand in the [NiFe] Center of the Regulatory H2-Sensing Hydrogenase from Ralstonia eutropha in Its Reduced State by HYSCORE and ENDOR Spectroscopy.
J. Am. Chem. Soc. 2003, 125, 13075–13083.
doi:
10.1021/ja036624x
[h2ases_2003a]
S. Reissmann, E. Hochleitner, H. Wang, A. Paschos, F. Lottspeich, R. S. Glass, A. Böck:
Taming of a Poison: Biosynthesis of the NiFe-Hydrogenase Cyanide Ligands.
Science 2003, 1067–1070.
doi:
10.1126/science.1080972
[h2ases_2002]
D. J. Evans, C. J. Pickett:
Chemistry and the hydrogenases.
Chem. Soc. Rev. 2003, 32, 268–275.
doi:
10.1039/b201317g
Mikroorganismen sind in der Lage, Stickstoff aus der Luft zur Stufe des Ammoniaks zu reduzieren und so in die Biosphäre einzuschleusen. Bakterien der Gattung Rhizobium leben mit Pflanzen aus der Familie der Fabaceae in Symbiose, woraus sich die seit langem genutzte Fähigkeit von Schmetterlingsblütlern wie Erbsen oder Lupinen ergibt, den Boden mit Stickstoff anzureichern.
Das stickstoffreduzierende bakterielle Enzym ist die Nitrogenase. In Abwesenheit von Stickstoff wirkt Nitrogenase als Hydrogenase, in Anwesenheit von Stickstoff wird die folgende Reaktion katalysiert:
N2 + 8 H+ + 8 e− = 2 NH3 + H2
Die Energie für diese endotherme Reaktion wird durch die Hydrolyse von 16 MgATP aufgebracht.
Nitrogenase besteht aus zwei verschiedenen Proteinen, dem Eisenprotein, das einen [4Fe-4S]-Cluster enthält, und dem MoFe-Protein. Das Fe-Protein koppelt nur zur Übertragung eines Elektrons an das MoFe-Protein. Dieses kumuliert die angelieferten Elektronen bis zu einem gewissen Reduktionsgrad. Steht beim Erreichen dieser Stufe N2 zur Verfügung, so beginnt die wahrscheinlich schrittweise Reduktion. Ist kein Stickstoff vorhanden, werden die angesammelten Elektronen auf H+ übertragen und es entsteht Wasserstoff.
Es liegt eine hochaufgelöste Strukturanalyse des MoFe-Proteins vor (1.16 Å Auflösung, PDB-Code: 1M1N):
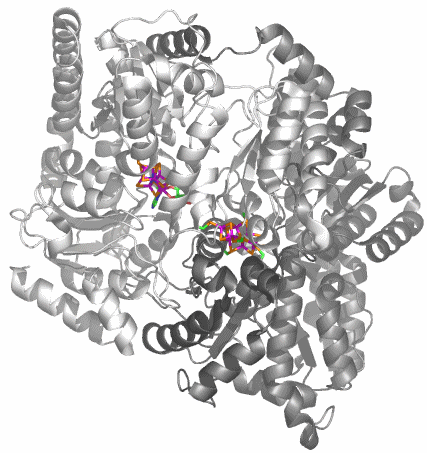
In dem aus zwei Proteinketten aufgebauten Enzym sind zwei metallhaltige Cofaktoren mit einzigartiger Struktur enthalten. Der „P-Cluster“ ist ein [8Fe-7S]-Eisenschwefelcluster, der von 4 Cys-Liganden in das Protein eingebunden ist. Ungewöhnlich ist der zentrale, an sechs Eisenatome bindende μ6-Sulfido-Ligand:
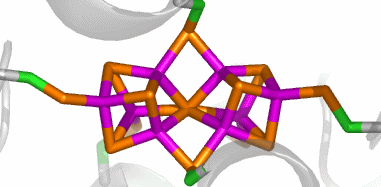
Als Funktion des P-Clusters wird die Akkumulation von Reduktionsäquivalenten gesehen. Der Ort der Stickstoffreduktion scheint ein zweiter ungewöhnlicher Eisen-Schwefel-Cluster zu sein, der MoFe-Cluster (im Bild des Holoenzyms das Metallzentrum links oben):
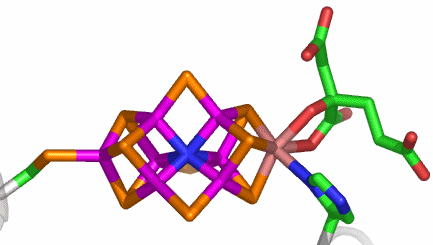
Der MoFe-Cofaktor ist ein ebenfalls ungewöhnlicher und in keinem anderen Enzym vorkommender [7Fe-9S]-X-Mo-Homocitrat-Cluster. „X“ wird in allen aktuellen Arbeiten als μ6-Nitrido-Ligand interpretiert. Der Cluster wird (im Bild rechts) von einem Molybdänatom komplettiert, das in oktaedrischer Koordination von 3 Sulfido-Liganden, einem zweizähnigen Homocitrato- und einem His-Ligand umgeben ist. Der gesamte Cofaktor, der zum Beispiel in N,N-Dimethylformamid löslich ist, wird von nur zwei Aminosäuren im Protein verankert, einem Fe-gebundenen Cys und einem Mo-bindenden His.
Die Kristallisation von N2-bindendem Protein ist bislang nicht gelungen, so dass die Formulierung eines Katalysecyclus spekulativ ist. Eine erste Schwierigkeit ist das Fehlen einer offensichtlichen Koordinationslücke. Man beachte dies, wenn in Vorschlägen zum Katalysecyclus an FeS4-Baueinheiten weitere Liganden gebunden werden [n2ase_2010], oder wenn Molybdän-zentrierte Reaktionskaskaden von Modellverbindungen auf den Nitrogenase-Cofaktor übertragen werden [n2ase_2009]. Eine Übersicht über die Koordinationschemie des N2-Liganden hilft, die spekulativen Aussagen zu Nitrogenase einzuordnen [n2ase_2004]. Entsprechend der Bedeutung des Enzyms interessiert auch hier die Cofaktor-Biosynthese und der Einbau in das Apoprotein [n2ase_2004a].
[n2ase_2010]
I. Dance:
Mimicking nitrogenase.
Dalton Trans. 2010, 39, 2972–2983.
doi:
10.1039/b922606k
[n2ase_2009]
B. M. Hoffman, D. R. Dean, L. C. Seefeldt:
Climbing Nitrogenase: Toward a Mechanism of Enzymatic Nitrogen Fixation.
Acc. Chem. Res. 2009, 42, 609–619.
doi:
10.1021/ar8002128
[n2ase_2004]
B. A. MacKay, M. D. Fryzuk:
Dinitrogen Coordination Chemistry: On the Biomimetic Borderlands.
Chem. Rev. 2004, 104, 385–401.
doi:
10.1021/cr020610c
[n2ase_2004a]
P. C. Dos Santos, D. R. Dean, Y. Hu, M. W. Ribbe:
Formation and Insertion of the Nitrogenase Iron-Molybdenum Cofactor.
Chem. Rev. 2004a, 104, 1159–1173.
doi:
10.1021/cr020608l
Cobalamin-abhängige Methionin-Synthase methyliert in Bakterien und Säugern Homocystein zu Met. Das Enzym überträgt dabei eine Methylgruppe von Methyl-tetrahydrofolat auf einen Cobalamin-Cofaktor, dieser S-methyliert anschließend das Thiol Homocystein. Eine eventuelle Nebenreaktion unter Bildung des inaktiven Cobalt(II)-Cofaktors kann in einem Nebenweg durch Reduktion und Reaktion mit Adenosylmethionin (AdoMet) repariert werden. Diese Chemie mit vier sterisch aufwändigen Substraten verlangt eine ungewöhnliche Beweglichkeit des Proteins, die Gegenstand aktueller Forschung ist.
PDB-Code 1K7Y bezieht sich auf die reduzierte Form des E.-coli-Enzyms (Auflösung: 3 Å). Der Cobalamin-Cofaktor ist nicht-kovalent und nicht-koordinativ in das Apoprotein eingelagert:
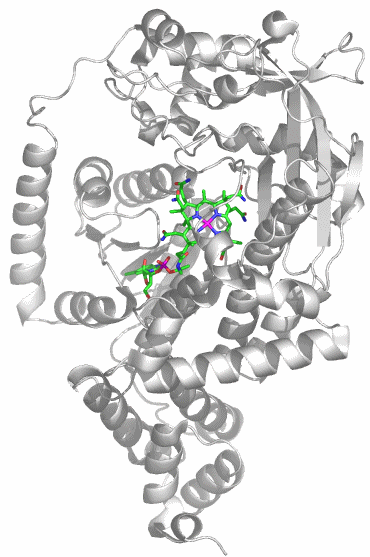
Der Cofaktor enthält Cobalt in der Oxidationsstufe +I. Das Zentralmetall ist als quadratisch-planar koordiniertes d8-Zentrum koordinativ gesättigt:
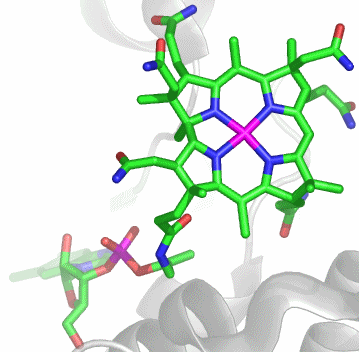
PDB-Code 1BMT zeigt mit 3 Å Auflösung das Enzym mit der methylierten Form des Cofaktors:
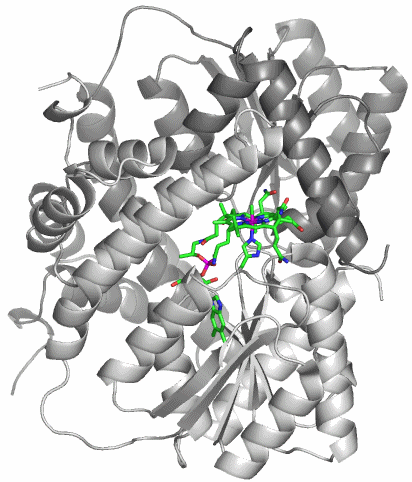
Die Oxidationsstufe des Zentralmetalls ist hier +III mit der Koordinationszahl 6, die jedoch auch hier nicht unter Beteiligung der Benzimidazol-Seitenkette zustande kommt, sondern durch die koordinative Bindung an eine His-Seitenkette des Proteins (vgl. die Formel in Fig. 1 von [cbl_2005a]:
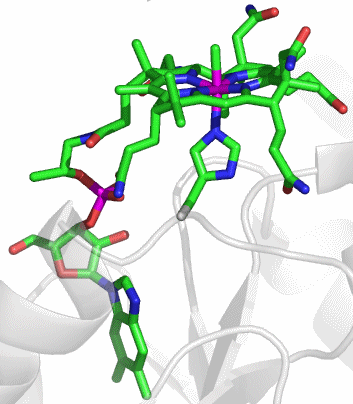
Ein Katalysecyclus ist in Scheme 21 (p. 2136) von [cbl_2005], der AdoMet-Seitenweg ist in Fig. 1 von [cbl_2004] mit aufgenommen. Die transferierten Methylgruppen werden jeweils als formale CH3+-Fragmente übertragen. Dies wird möglich, da das quadratisch-planare low-spin d8-Cobalt(I)-Zentrum als Metallbase wirkt und über ein Elektronenpaar im d(z2)-Orbital als Nucleophil an Methyl-tetrahydrofolat angreifen kann, um anschließend als Abgangsgruppe das Carbenium-Fragment einem Thiolat zu überlassen. Durch die Einstufung einer metallgebundenen Methylgruppe als CH3−-Ligand wird diese Reaktionsfolge zu einem 2-e-Redoxprozess am Zentralmetall:
Zahlreiche Anaerobier weisen einen CO-Stoffwechsel auf, indem sie CO zu CO2 oxidieren und Reduktionsäquivalente bei sehr niedrigem Potential (E0' = −0.56 V) gewinnen oder indem sie CO aus CO2 als Kohlenstoffbaustein herstellen [codh_2004a]. Das Leben unter CO wird vor allem durch zwei Enzymgruppen ermöglicht, deren Strukturen kürzlich aufgeklärt wurden: Kohlenmonoxid-Dehydrogenasen (CODHs) und Acetylcoenzym-A-Synthasen (ACSs). In diesem Kapitel wird auf eine der beiden CODHs eingegangen, auf NiFe-CODH (eine Mo-CODH wird nicht behandelt), ferner auf den ACS-Teil einer bifunktionellen CODH/ACS.
Kohlenmonoxid-Dehydrogenase/Acetylcoenzym-A-Synthase (CODH/ACS) ist ein bifunktionelles Enzym, dass in acetogenen, methanogenen oder sulfatreduzierenden Anaerobiern, die unter CO2 als einziger Kohlenstoffquelle wachsen können, CO2 zu CO reduziert und dieses anschließend zusammen mit einer Methylgruppe unter Bildung von Acetyl-CoA an Coenzym A bindet. Die CO-Dehydrogenase-Funktion ist im sogenannten C-Cluster des Enzyms lokalisiert, die ACS-Aktivität im A-Cluster. Die am C-Cluster katalysierte Reaktion ist:
CO2 + 2 H+ + 2 e− = CO + H2O
Das gebildete CO gelangt – ohne das Enzym zu verlassen – durch einen Kanal zum A-Cluster, wo es mit zwei Reaktionspartnern gekoppelt wird: mit einer Methylgruppe aus einem Methylcobalamin-Eisen-Schwefel-Protein (engl. corrinoid iron sulfur protein, CFeSP) und mit Coenzym A:
CH3-CoIII-CFeSP + CO + CoA-SH = CoI-CFeSP + CoA-S-C(O)CH3 + H+
(Man beachte die hier unglücklich zusammentreffenden üblichen Kürzel: Co bedeutet zum einen Cobalt, zum anderen Coenzym.)
Es liegt eine Strukturanalyse an Moorella-thermoacetica-CODH/ACS vor (2.2 Å Auflösung, PDB-Code 1MJG [codh_2002]. Dargestellt ist eine α- und eine β-Kette des α2β2-Heterotetramers. Die beiden Reaktionszentren, der A-Cluster (oben) und der C-Cluster (unten) sind durch einen hydrophoben Kanal verbunden, durch den CO diffundieren kann.
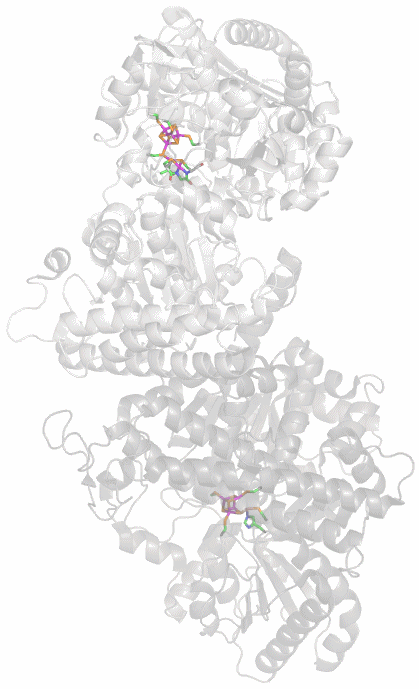
Das aktive Zentrum der β-Kette ist der C-Cluster, ein [4Fe-Ni-3S]-Fragment, das von drei Fe-Cys-Kontakten sowie einer Ni-Cys und einer Ni-His-Bindung in das Protein eingebunden ist. Dasselbe Strukturelement wird auch in CODHs methanogener Bakterien gefunden [codh_2008]. Der kürzeste Fe-Ni-Abstand beträgt 2.67 Å:
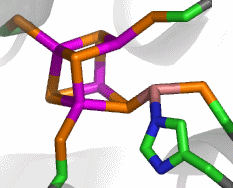
Eine Metall-Kohlenstoff-Bindung wird im Katalysecyclus am aktiven Zentrum der α-Kette formuliert. In diesem A-Cluster ist ein [4Fe-4S]-Baustein über einen verbrückenden Cys-Ligand mit einer binuklearen Einheit verbunden. Deren Aufbau ist ebenso ungewöhnlich wie umstritten. Das linke, hell-türkisblau gezeichnete Metallatom bindet einen Acetyl-Ligand, dessen Identität bei einer Auflösung von 2.2 Å als unsicher betrachtet werden darf. Auch die Identität des Metallions ist nicht völlig klar. Bei Beugungsversuchen mit hoher anomaler Dispersion der einzelnen vermuteten Metallatome ergibt sich die bevorzugte Besetzung mit Kupfer. Eine zweite Strukturanalyse kommt dagegen zu dem Ergebnis, dass zumindest in der katalytisch aktiven Form [codh_2004] ein zweites Nickelatom diese Position besetzt.
Das rechte Metallatom wird übereinstimmend als Nickel identifiziert. Die quadratisch-planare Umgebung ist für Nickel(II) nicht ungewöhnlich, die Ligandausstattung schon. Neben zwei verbrückenden Cys-Liganden ist der seltene Fall realisiert, dass die Proteinhauptkette Ligandatome zur Verfügung stellt, und zwar die wohl deprotonierten Amid-N-Atome der Cys-Gly-Cys-Einheit, die auch die beiden μ-SCys-Liganden zur Verfügung stellt.
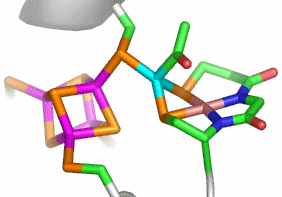
So ungewöhnlich der Aufbau des A-Clusters ist, so umstritten ist er derzeit auch. Es liegt eine zweite Strukturanalyse an CODH/ACS desselben Organismus vor, die zu einem anderen Schluss kommt (1.9 Å Auflösung, PDB-Code 1OAO). Neben einem A-Cluster in einer „geschlossenen“ Konformation – diese Form wird in 1MJG ausschließlich gefunden –, der anstelle des Kupferatoms ein Zinkatom aufweist, wird ein zweiter A-Cluster in einer reaktionsbereiten „offenen“ Konformation gefunden. Die Kupfer/Zink-Lage ist hier von Nickel belegt:
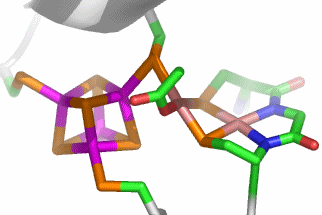
Zugleich werden Ergebnisse vorgelegt, die das Vorhandensein dieser Ni2-Form als notwendige Voraussetzung für die Aktivität des Enzyms aufzeigen.
Der Ablauf der Katalyse ist weitgehend spekulativ. Fig. 3 in [codh_2002] zeigt einen Vorschlag, bei dem das durch vier anionische Liganden elektronenreiche Nickel(II)-Zentrum ein CH3+-Fragment von Methylcobalamin übernimmt – es bildet sich eine Metall-Methyl-Bindung. Bei der anschließenden Kopplung des an Kupfer(I) vermuteten Carbonyl-Liganden mit der Methylgruppe sind die durchlaufenen elektronischen Zustände völlig unklar.
Alternativ wird von Nickel(0) als das dem Eisen-Schwefel-Cluster benachbarte Metall ausgegangen. An dieses wird CO koordiniert, CH3+ oxidativ addiert und anschließend der Acetyl-Ligand durch CO-Insertion in die Metall-Methyl-Bindung gebildet. Diese Sichtweise entspricht völlig dem Ablauf des Monsanto-Essigsäure-Prozesses (Fig. 5 in [codh_2003]. Einen Überblick aus der Sicht organometallchemischer Modellreaktion gibt [codh_2005].
Bei der Klausur … : Alkyl-Metall-Komplexe als Alkylierungsmittel; Stellung kovalent bindender Liganden in der spektrochemischen Reihe; Alkylübertragung als oxidative Addition/reduktive Eliminierung.
[cbl_2005]
K. L. Brown:
Chemistry and Enzymology of Vitamin B12.
Chem. Rev. 2005, 105, 2075–2149.
doi:
10.1021/cr030720z
[cbl_2005a]
K. P. Jensen:
Electronic Structure of Cob(I)alamin: The Story of an Unusual Nucleophile.
J. Phys. Chem. B 2005, 109, 10505–10512.
doi:
10.1021/jp050802m
[cbl_2004]
J. C. Evans, D. P. Huddler, M. T. Hilgers, G. Romanchuk, R. G. Matthews, M. L. Ludwig:
Structures of the N-terminal modules imply large domain motions during catalysis by methionine synthase.
PNAS 2004, 101, 3729–3736.
doi:
10.1073/pnas.0308082100
Noch berücksichtigen:
[cbl_2011]
S.-L. Chen, M. R. A. Blomberg, P. E. M. Siegbahn:
How Is a Co-Methyl Intermediate Formed in the Reaction of Cobalamin-Dependent Methionine Synthase? Theoretical Evidence for a Two-Step Methyl Cation Transfer Mechanism.
The Journal of Physical Chemistry B 2011, 115, 4066–4077.
doi:
10.1021/jp105729e
[codh_2011]
Y. Kung, C. L. Drennan:
A role for nickel-iron cofactors in biological carbon monoxide and carbon dioxide utilization.
Curr. Opin. Chem. Biol. 2011, 15, 276–283.
doi:
10.1016/j.cbpa.2010.11.005
[codh_2009]
Y. Kung, T. I. Doukov, J. Seravalli, S. W. Ragsdale, C. L. Drennan:
Crystallographic Snapshots of Cyanide- and Water-Bound C-Clusters from Bifunctional Carbon Monoxide Dehydrogenase/Acetyl-CoA Synthase.
Biochemistry 2009, 48, 7432–7440.
doi:
10.1021/bi900574h
[codh_2008]
W. Gong, B. Hao, Z. Wei, D. J. Ferguson, Jr., T. Tallant, J. A. Krzycki, M. K. Chan:
Structure of the α2ε2 Ni-dependent CO dehydrogenase component of the Methanosarcina barkeri acetyl-CoA decarbonylase/synthase complex.
PNAS 2008, 105, 9558–9563.
doi:
10.1073/pnas.0800415105
[codh_2005]
T. C. Harrop, P. K. Mascharak:
Structural and spectroscopic models of the A-cluster of acetyl coenzyme a synthase/carbon monoxide dehydrogenase: Nature’s Monsanto acetic acid catalyst.
Coord. Chem. Rev. 2005, 249, 3007–3024.
doi:
10.1016/j.ccr.2005.04.019
[codh_2004]
J. Seravalli, Y. Xiao, W. Gu, S. P. Cramer, W. E. Antholine, V. Krymov, G. J. Gerfen, S. W. Ragsdale:
Evidence That NiNi Acetyl-CoA Synthase Is Active and That the CuNi Enzyme Is Not.
Biochemistry 2004, 43, 3944–3955.
doi:
10.1021/bi036194n
[codh_2004a]
S. W. Ragsdale:
Life with Carbon Monoxide.
Critical Reviews in Biochemistry and Molecular Biology 2004a, 39, 165–195.
doi:
10.1080/10409230490496577
[codh_2003]
C. Damault, A. Volbeda, E. J. Kim, P. Legrand, X. Vernède, P. A. Lindahl, J. C. Fonticella-Camps:
Ni-Zn-Fe4-S4] and Ni-Ni-Fe4-S4] clusters in closed and open α subunits of acetyl-CoA synthase/carbon monoxide dehydrogenase.
Nat. Struct. Biol. 2003, 10, 271–279.
doi:
10.1038/nsb912
[codh_2002]
T. I. Doukov, T. M. Iverson, J. Seravalli, S. W. Ragsdale, C. L. Drennan:
A Ni-Fe-Cu Center in a Bifunctional Carbon Monoxide Dehydrogenase/Acetyl-CoA Synthase.
Science 2002, 298, 567–572.
doi:
10.1126/science.1075843
Angesichts der Fülle von Enzymen ist es erstaunlich, dass nur eine Handvoll davon in isolierter Form großtechnische Verwendung finden. Unter den Enzymen mit Metall-Zentrum ist dies die Xylose-Isomerase, die im Tonnen-Maßstab hergestellt und für die Isomerisierung von Glucose zur Glucose/Fructose-Gleichgewichtsmischung (“high fructose syrup”) eingesetzt wird – in Nordamerika das wichtigste Süßungsmittel weit vor der bei uns bedeutenderen Saccharose (Rohrzucker, Rübenzucker). Xylose-Isomerase ist ein metallabhängiges Enzym mit zweikernigem aktivem Zentrum, in dem eine Hydridverschiebung an der zur offenkettigen Form aufgefalteten Glucose die isomere Ketose erzeugt. Xylose-Isomerase ist wohl das schönste Beispiel für ein Enzym, das trotz enormer wirtschaftlicher Bedeutung auch nicht ansatzweise modelliert worden ist – eine lohnende Aufgabe schon wegen der Möglichkeit, das Enzym durch einen kostengünstigeren synthetischen, „biomimetischen“, Katalysator zu ersetzen. Die katalytisch aktiven Metalle im Enzym sind übrigens austauschbar: Mangan(II) in der Natur, in der Technik auch Magnesium und Cobalt(II).
Es liegen hochaufgelöste Strukturanalysen für Xylit- und Glucose-beladene Xylose-Isomerase vor (Auflösung < 1 Å) [xyl_2004]. Das Holoenzym ist in der mit Xylit beladenen Form gezeigt (PDB-Code 1S5N):
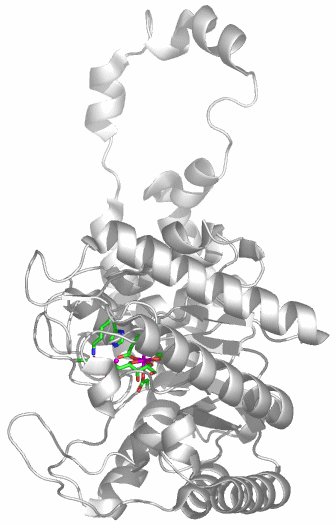
Das aktive Zentrum ist ungewöhnlich hoch hydratisiert (gepunktete Kugeln). Ein Xylit-Molekül bindet mit O2 und O4 unter Ausbildung eines Chelat-Sechsrings an die vollbesetzte rechte Manganposition. Die weiteren Liganden des rechten Manganatoms sind vier Carboxylate von Asp- und Glu-Seitenketten. Das zweite Manganatom ist über drei Lagen fehlgeordnet. Dessen weitere Liganden sind drei Asp/Glu-Reste, ein His, sowie Wassermoleküle. Xylit hat bindende Kontakte außer zu den Metallatomen und Wassermolekülen auch zu einem Lys-Rest im linken Bildteil.
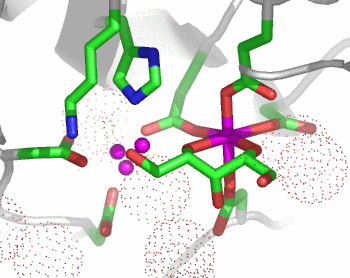
Beim Eintauchen der Kristalle in Glucoselösungen wird ein α-Glucose-Addukt erhalten, in dem das linke Manganatom die äußerst linke Lage der vorigen Abbildung einnimmt und in dem die Aldose mit O3 und O4 einen Chelatfünfring mit dem rechten Manganatom aufbaut (PDB-Code 1S5M):
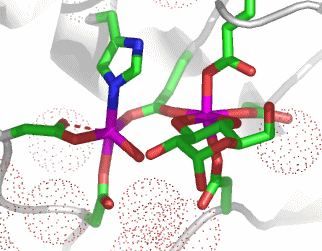
Es ist überraschend, den α-Glucopyranose-Ligand vorzufinden, der im wässrigen Lösungsgleichgewicht nur zu einem Drittel vorliegt; die Hauptspezies ist vielmehr das β-Anomer. Im folgenden Bild sind Wechselwirkungen dargestellt, die wohl zur Verschiebung des Anomerengleichgewichts beitragen: (1) zwei Wassermoleküle bilden Wasserstoffbrückenbindungen zum α-O1; (2) die hydrophobe Seitenkette eines Phenylalanins die Annäherung von Wassermolekülen an ein β-O1 verhindern, vielleicht sogar die Positionierung der β-Hydroxylgruppe selbst. Die Spezifität für Xylose und Glucose scheint durch die weitere Auskleidung des aktiven Zentrums gesichert zu werden: zwei Tryptophan-Seitenketten bilden ein hydrophobes Dach und einen Boden, der Äquatorbereich des Pyranoserings ist jedoch hydrophil. Zucker mit axialen Hydroxylgruppen sollten durch diese Gestaltung des Raums benachteiligt sein.
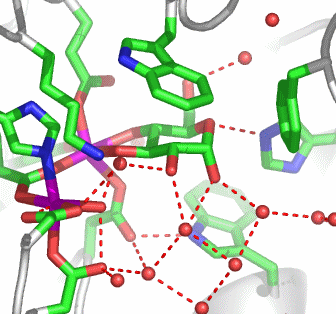
Dasselbe Ergebnis wird bei einer kombinierten Röntgen- (PDB-Code 3KBM) und Neutronenbeugungsuntersuchung (PDB-Code 3KCL) an einer Cd2-Variante des Enzyms erhalten (Auflösung: 2 Å).
Durch dieselbe Methodenkopmbination, nun mit dem katalytisch wirksamen Magnesium im aktiven Zentrum, wird der Zucker im produktbeladenen Enzym als offenkettige keto-Xylulose gefunden (PDB-Code 3CWH):
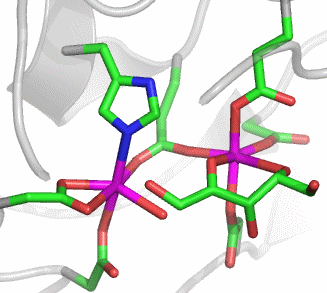
Eines der beiden Magnesium-Atome ist nun Teil eines Chelatsechsrings mit den Xylulose-O-Atomen O2 und O4 [xyl_2008].
Dasselbe Bindungsmotiv wird gefunden – wieder durch kombinierte Röntgen- und Neutronenbeugung –, wenn die katalytisch aktiven Metallionen durch inhibierende Nickel(II)-Ionen ersetzt werden und die Kristalle mit Glucose durchtränkt werden. Die Röntgen- (PDB-Code 3KBN) und Neutronen-Analyse (PDB-Code 3KCO) zeigen nun (bei 1.53 Å Auflösung) einen Glucose-Liganden, der als offenkettiges aldehydo-Isomer an die (inhibierenden) Nickelzentren gebunden ist (links: C1, rechts: C6):
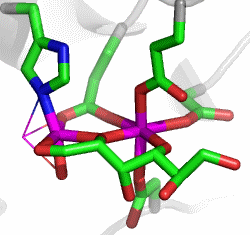
Die linke Nickellage zeigt Fehlordnung. Das gezeigte Nickelatom ist nur zu etwa der Hälfte besetzt, ebenso wie eine noch etwas weiter links liegende Nickellage, deren Koordination durch den Histidin-, Aqua/Hydroxido und Glutamat-Ligand durch dünne Striche angedeutet ist [xyl_2010]. Anders als beim Produktkomplex liegt beim inhibierten Nickel-Edukt-Komplex ein zweiter Chelatring mit O1 und O2 vor.
Für die Aldose/Ketose-Epimerisierung ist Säure/Base-Katalyse zu erwarten. Vor diesem Hintergrund überrascht die Auskleidung des aktiven Zentrums. Mangan(II) ist ebenso wie das als Ersatz geeignete Magnesium ohnehin eine sehr schwache Lewis-Säure. Insgesamt sechs Carboxylat-Funktionen und nur ein His-Ligand sollte keine für die Katalyse verfügbare Acidität mehr übrig lassen. Es fällt auf, dass bei der Bindung von α-Glucose die reagierenden Funktionen an C1 und C2 sich nicht unmittelbar am Metall befinden. Es könnte daher in Betracht gezogen werden, dass in diesem Beispiel die Metallatome nur der Positionierung des Substrats dienen könnten ohne in die Reaktion selbst einzugreifen.
Eine minimale Version einer sauer katalysierten Isomerisierung ist für α-Xylose als Substrat im folgenden Schema dargestellt; in ähnlicher Weise wird auch für metallfreie Epimerasen formuliert:
Schritt a ist die säurekatalysierte Ringöffnung. Das Carbenium-Ion stabilisiert sich in Schritt b entweder durch Hydridverschiebung oder durch Deprotonierung/Endiol-Bildung/Reprotonierung. In Schritt c kommt es zum Ringschluss durch O5, wobei das zur Katalyse benötigte H+ wieder abgespalten wird. Das oben gezeigte Bild vom aktiven Zentrum des Enzyms zeigt eine strukturelle Voraussetzung für diesen Ablauf, nämlich ein His in H-Brückenentfernung zum Ring-O-Atom.
Die Autoren der Neutroneneugungsuntersuchung formulieren hinsichtlich der Protonierungs-/Deprotonierungsschritte erstaunlicherweise ähnlich, die sonst übliche Bereitstellung eines Hydroxid-Nukleophils wird nicht in Betracht gezogen, sondern ein Aqua-Ligand wird als Säure gesehen. In diesem Zusammenhang ist es erstaunlich, dass ein unerwartetes Protonierungsmuster an den reduzierten Termini von Xylulose nd Glucose gefunden wird (O5 der offenkettigen Formen soll deprotoniert und Akzeptor in einer Wasserstoffbrückenbindung zu einer HisH+-Funktion sein). Die Funktionsweise der Xylose-Isomerase ist also bei weitem noch nicht geklärt.
[xyl_2010]
A. Y. Kovalevsky, L. Hanson, S. Z. Fisher, M. Mustyakimov, S. A. Mason, V. Trevor Forsyth, M. P. Blakeley, D. A. Keen, T. Wagner, H. L. Carrell, A. K. Katz, J. P. Glusker, P. Langan:
Metal Ion Roles and the Movement of Hydrogen during Reaction Catalyzed by d-Xylose Isomerase: A Joint X-Ray and Neutron Diffraction Study.
Structure 2010, 18, 688–699.
doi:
10.1016/j.str.2010.03.011.
[xyl_2008]
A. Y. Kovalevsky, A. K. Katz, H. L. Carrell, L. Hanson, M. Mustyakimov, S. Z. Fisher, L. Coates, B. P. Schoenborn, G. J. Bunick, J. P. Glusker, P. Langan:
Hydrogen Location in Stages of an Enzyme-Catalyzed Reaction: Time-of-Flight Neutron Structure of d-Xylose Isomerase with Bound d-Xylulose.
Biochemistry 2008, 47, 7595–7597.
doi:
10.1021/bi8005434
[xyl_2004]
T. D. Fenn, D. Ringe, G. A. Petsko:
Xylose Isomerase in Substrate and Inhibitor Michaelis States: Atomic Resolution Studies of a Metal-Mediated Hydride Shift.
Biochemistry 2004, 43, 6464–6474.
doi:
10.1021/bi049812o
Übersicht über die Klausurstichwörter:
Allgemeine Koordinationschemie und Säure-Base-Chemie:
Faustregeln kennen, um die Stabilität von Komplexen abzuschätzen; LFSE-Werte ausrechnen; Jahn-Teller-Verzerrung.
Kristallfeldaufspaltung beim Oktaeder und Tetraeder und bei verzerrten Formen, Auswahlregeln in Elektronenspektren, charge-transfer-Übergänge.
Selbstorganisation, Mustererkennung, Spinkopplung in Clustern mit Brückenliganden, Superaustausch.
Koordinationsgeometrie bei verschiedenen dn-Konfigurationen; formale und physikalische (spektroskopische) Oxidationsstufe; Lage eines spintragenden Orbitals im Kristallfeld.
Aufbau und Acidität von Aqua-Metall-Komplexen; Faktoren, welche die Acidität von metallgebundenen Aqua-Liganden beeinflussen (Koordinationszahl, Ladungsdichte am Zentralmetall, Einbindung der Protonen in Wasserstoffbrückenbindungen).
Einschränkung der Koordinationsgeometrie durch Ligandfeldstabilisierungsenergie (LFSE); Darstellung einer Inhibitorwirkung im Energieschema einer Reaktion; Acidität und Nukleophilie verbrückender Aqua/Hydroxido-Liganden.
Redox-Chemie:
Umgang mit Potentialdiagrammen, Erkennen der freiwillig ablaufenden Redoxreaktion.
Einfluss von Liganden auf elektrochemische Potentiale, Bedeutung von Potentiallagen in Reaktionskaskaden, Reorganisationsenergie.
pH-Abhängigkeit elektrochemischer Potentiale, Stabilisierung hoher/niedriger Oxidationsstufen.
Bedeutung der Elektrostatik bei der Abschätzung des Redoxpotentials; Stabilisierung hoher Oxidationsstufen durch anionische Liganden.
Zusammenhang zwischen der Energie der Metall-Grenzorbitale und der Redox-Reaktivität; Abhängigkeit der Grenzorbitalenergie von der Ligandanordnung.
Spinzustände, Starkfeldliganden:
Voraussetzungen für den high-spin–low-spin-Übergang; Auswirkung des Übergangs auf die Metall-Ligand-Bindungsordnung und die Kovalenz der Bindung; Aufspaltung der d-Niveaus in Porphyrin-Komplexen, räumliche Lage der Grenzorbitale in geläufigen zweiatomigen Liganden.
Unterschiede zwischen wichtigen zweiatomigen Liganden mit deren Grenzorbitalen verknüpfen: CO, CN−, NO+, NO, NO−, O2, O2•−, O22−.
Stellung von π-Säuren in der spektrochemischen Reihe; Darstellung der π-Säure–Zentralmetall-Wechselwirkung im Orbitalbild.
Alkyl-Metall-Komplexe als Alkylierungsmittel; Stellung kovalent bindender Liganden in der spektrochemischen Reihe; Alkylübertragung als oxidative Addition/reduktive Eliminierung.
Die angegebenen Ionenradien sind unkorrigierte Shannon-Prewitt-Radien nach R. D. Shannon: Crystal Radii in Oxides and Fluorides, in: Encyclopedia of Inorganic Chemistry, Vol. 2 (Ed. R. B. King), p. 929–942, Wiley 1994.
Die pKA-Werte der hydratisierten Metall-Kationen sind entnommen aus: D. T. Richens: The Chemistry of Aqua Ions, Wiley 1997.
| Ionenradius/Å[Koord.] | 0.77[hs-4t], 0.78[hs-4sp], 0.92[hs-6o], 0.75[ls-6o] |
| Konfiguration | d6 |
| LFSE[6o]/Dq | hs: −4, ls: −24 |
| bevorzugte Koordination | hs: (N,O)5–6, S4, ls: N6, N5(O,S) |
| Lösungsspezies bei pH 7 | [Fe(H2O)5–6]2+ |
| pKA des Aqua-Ions | 9.5 |
| + NH3 | kaum [Fe(NH3)4–6]2+, Fe(OH)2 |
| + H2S zur Metallsalz/NH3-Lösung | FeS |
| + CN− | [Fe(CN)6]4− |
| Ionenradius/Å[Koord.] | 0.63[hs-4t], 0.785[hs-6o], 0.69[ls-6o] |
| Konfiguration | d5 |
| LFSE/Dq | hs: 0, [ls-6o]: −20 |
| bevorzugte Koordination | hs: O6, ls: (N,O)6 |
| Spezies bei pH 7 | Fe(OH)3 |
| pKA des Aqua-Ions | 2.2 |
| + NH3 | Fe(OH)3 |
| + H2S zur Metallsalz/NH3-Lösung | Fe(OH)3 |
| + CN− | [Fe(CN)6]3− |
| Ionenradius/Å[Koord.] | 0.74[4t], 0.91[6o] |
| Konfiguration | d10 |
| LFSE/Dq | 0 |
| bevorzugte Koordination | N4, S4 |
| Lösung bei pH 7 | Disproportionierung |
| pKA des Aqua-Ions | – |
| E0' | 0.1 V |
| + NH3 | [Cu(NH3)4]+ |
| + H2S zur Metallsalz/NH3-Lösung | Cu2S |
| + CN− | [Cu(CN)4]3− |
| Ionenradius/Å[Koord.] | 0.71[4], 0.79[5], 0.87[6o] |
| Konfiguration | d9 |
| LFSE/Dq | > 0 |
| bevorzugte Koordination | N/O4–6, JT-verzerrt |
| Lösung bei pH 7 | [Cu(H2O)5]2+ vgl. [Cu1] |
| pKA des Aqua-Ions | 8.0 |
| E0' | 0.1 V |
| + NH3 | [Cu(NH3)4(H2O)2]2+ |
| + H2S zur Metallsalz/NH3-Lösung | „CuS“, enthält neben CuII und S2− auch CuI und S22− |
| + CN− | Reduktion zu [Cu(CN)4]3− |
| Ionenradius/Å[Koord.] | 0.97[hs-6o] |
| Konfiguration | d5 |
| LFSE/Dq | 0 |
| bevorzugte Koordination | (N,O)6 |
| Spezies bei pH 7 | [Mn(H2O)6]2+ |
| pKA des Aqua-Ions | 10.6 |
| + NH3 | Mn(OH)2 |
| + H2S zur Metallsalz/NH3-Lösung | MnS |
| + CN− | ls-[Mn(CN)6]4− |
| Ionenradius/Å[Koord.] | 0.785[hs-6] |
| Konfiguration | d4 |
| LFSE/Dq | > 0 |
| bevorzugte Koordination | O4–6, JT-verzerrt |
| Spezies bei pH 7 | Mn(OH)3; MnOOH |
| pKA des Aqua-Ions | 0.7 |
| + NH3 | Mn(OH)3, MnOOH?? |
| + H2S zur Metallsalz/NH3-Lösung | ? |
| + CN− | ls-[Mn(CN)6]3−?? |
| Ionenradius/Å[Koord.] | 0.67[6o] |
| Konfiguration | d3 |
| LFSE/Dq | −12 |
| bevorzugte Koordination | O6 |
| Spezies bei pH 7 | MnO2 |
| pKA des Aqua-Ions | – |
| + NH3 | –?? |
| + H2S zur Metallsalz/NH3-Lösung | ? |
| + CN− | –?? |
| Ionenradius/Å[Koord.] | 0.69[4], 0.83[6o] |
| Konfiguration | d8 |
| LFSE[6o]/Dq | −12 |
| bevorzugte Koordination | (N,O)6 |
| Lösung bei pH 7 | [Ni(H2O)6]2+ |
| pKA des Aqua-Ions | 9.9 |
| + NH3 | [Ni(NH3)6]2+ |
| + H2S zur Metallsalz/NH3-Lösung | NiS |
| + CN− | [Ni(CN)4]2−, [Ni(CN)5]3− |
| Ionenradius/Å[Koord.] | 0.74[4t], 0.82[5], 0.88[6o] |
| Konfiguration | d10 |
| LFSE/Dq | 0 |
| bevorzugte Koordination | N4, (N,O)5–6, S4 |
| Lösungsspezies bei pH 7 | [Zn(H2O)5–6]2+ |
| pKA des Aqua-Ions | 9.0 |
| + NH3 | [Zn(NH3)4]2+ |
| + H2S zur Metallsalz/NH3-Lösung | ZnS |
| + CN− | [Zn(CN)4]2− |
Die folgende Graphik zeigt elektrochemische Potentiale bei pH 7 und Aktivität 1. Die Zahlenwerte sind entnommen: D. M. Kurtz jr.: Dioxygen-binding Proteins. CCC 8, 229–260 (230).
Die Zahl der übertragenen Elektronen ist farblich codiert:
schwarz: 1 Elektron,
blau: 2 Elektronen,
grün: 3 Elektronen,
rot: 4 Elektronen.